概述
cDNA文庫是以特定的組織或細胞mRNA為模板,逆轉錄形成的互補DNA(cDNA)與適當的載體(常用噬菌體或質粒載體)連線後轉化受體菌形成重組DNA克隆群,這樣包含著細胞全部mRNA信息的cDNA克隆集合稱為該組織或細胞的cDNA文庫。cDNA文庫特異地反映某種組織或細胞中,在特定發育階段表達的蛋白質的編碼基因,因此cDNA文庫具有組織或細胞特異性。
cDNA文庫顯然比基因組DNA文庫小得多,能夠比較容易從中篩選克隆得到細胞特異表達的基因。但對真核細胞來說,從基因組DNA文庫獲得的基因與從cDNA文庫獲得的不同,基因組DNA文庫所含的是帶有內含子和外顯子的基因組基因,而從cDNA文庫中獲得的是已經過剪接、去除了內含子的cDNA。
真核生物基因組DNA十分龐大,其複雜程度是蛋白質和mRNA的100倍左右,而且含有大量的重複序列. 採用電泳分離和雜交的方法,都難以直接分離到目的基因.這是從染色體DNA為出發材料直接克隆目的基因的一個主要困難。
高等生物一般具有10^5種左右不同的基因,但在一定時間階段的單個細胞或個體中,都僅有15%左右的基因得以表達,產生約15000種不同的mRNA分子.可見,由mRNA出發的cDNA克隆,其複雜程度要比直接從基因組克隆簡單得多。
cDNA文庫在研究具體某類特定細胞中基因組的表達狀態及表達基因的功能鑑定方便具有特殊的優勢,從而使它在個體發育、細胞分化、細胞周期調控、細胞衰老和死亡調控等生命現象的研究中具有更為廣泛的套用價值,是研究工作中最常使用到的基因文庫。
定義
(cDNA Library)某種生物基因組轉錄的全部mRNA經反轉錄產生的cDNA片段分別與克隆載體重組,並將其引入到相應的宿主細胞中(一般為 E.coli.)繁殖和擴增,理論上此群體就包含有該物種的全部mRNA信息,稱該生物基因組的cDNA文庫。
原理
經典cDNA文庫構建的基本原理:用Oligo(dT)作逆轉錄引物,或者用隨機引物,給所合成的cDNA 加上適當的連線接頭,連線到適當的載體中獲得cDNA文庫。其基本步驟包括:(1)mRNA的提純獲取高質量的mRNA是構建高質量的cDNA文庫的關鍵步驟之一。(2)cDNA第一條鏈的合成。(3)cDNA第二條鏈的合成。(4)雙鏈cDNA的修飾。(5)雙鏈cDNA的分子克隆。(6)cDNA文庫的擴增。(7)cDNA文庫鑑定評價。
注意事項
構建全長cDNA文庫分為噬菌體文庫和質粒文庫,二者大同小異。無論怎樣,應當注意如下幾個方面:
獲得起始RNA
構建cDNA文庫要求的RNA量比做RACE和Northern blot的要多,在材料允許的情況下一般的試劑盒均推薦採用純化總mRNA後進行反轉錄,這比直接採用總RNA進行反轉錄而構建的cDNA文庫好,雖然後者也並不是不能做。老版本CLONTECH的SMART 4的中級柱子要求純化後的總mRNA量最好在0.05-0.5微克左右,這就要求起始總RNA量較多。雖然有的試劑盒聲稱少至幾十個納克的總RNA也可以構建cDNA文庫,但這是針對材料極為稀缺者而言,但起始RNA太少還是會或多或少影響文庫構建成功的風險和文庫的代表性。至於RNA的質量,如果採用純化總mRNA後反轉錄,則對總RNA的雜質方面要求稍松,但對RNA的完整性則一絲不苟,要求未降解。如果直接採用總RNA進行反轉錄,則對總RNA的質量要求非常高,不僅要求RNA相當完整而無降解,而且要求多酚、多糖、蛋白、鹽、異硫氰酸胍等雜質少,最好是試劑盒抽提的。
反轉錄
這是構建cDNA文庫中最貴的一步,也是核酸質變的一步,它將易降解的RNA變成了不易降解的cDNA。反轉錄不成功,說明一次文庫方案的夭折。反轉錄效率不高表現在一是部分mRNA被反轉錄了,但還有相當一部分本該反轉錄的mRNA未被反轉總的全長cDNA太少,這就難以構建好的全長cDNA文庫。少量程度的mRNA降解或反轉錄不完全在SMART 4等試劑盒及手工方法構建中對文庫的滴度影響不大,但對文庫的全長性則有很大影響。Invitrogen公司基於去磷酸化、去帽、RNA接頭連線後再反轉錄的新技術(可參考其GeneRacer說明書)從原理上是保證最終獲得全長cDNA的最好方法,但對mRNA的完整性要求非常高,理論上講必須是帶有帽子結構和Poly A結構的全長mRNA且反轉錄完全,才能進入文庫中。反轉錄完成後點樣檢測cDNA的濃度及分子量分布是很重要的。
層析柱cDNA分級
這一步稍不注意會影響成功性或影響獲得的cDNA的片段分布特點。這一步的操作要小心,尤其要在加入cDNA之前通過反覆懸浮和試滴保證柱子能正常工作,cDNA的加入和收集要精力集中。獲得的每一級的cDNA量很少,檢測時帶型很暗,所以要用新鮮做的透明薄膠檢測,根據檢測結果一定要捨棄太短的cDNA(一般400bp以下就不要了,因為短片段太多會嚴重影響後面的連線轉化效果及文庫質量)。
四、噬菌體文庫或質粒文庫均對載體與cDNA的連線效率要求很高,也對連線產物轉染或轉化大腸桿菌的效率要求很高。連線效率高低直接關係到文庫構建是否成功,更要注意的是文庫連線與一般的片段克隆的連線不一樣。一般的片段克隆連線是固定長度的載體與固定長度的目的DNA連線,而文庫連線是固定長度的載體與非固定長度的目的DNA連線,目的基因cDNA長的有10kb以上,短的只有500bp或更短。一系列長度不等的cDNA與載體在一起連線的結果,不同長度cDNA的連線效率就不一樣。有的專家的經驗是,根據分級結果,有意識地將長度不同的cDNA群分別與載體連線,再分別轉化或轉染大腸桿菌,分別完成滴度檢測,最後將不同長度級別的文庫混合在一起供雜交篩選。
mRNA
製備
動物細胞mRNA的製備
植物細胞mRNA的製備
來源
選用mRNA含量高的組織材料,或通過藥物等方法提高mRNA的含量
完整性
1、mRNA在無細胞翻譯體系指導合成高分子量蛋白質的能力。無細胞翻譯系統(Cell-free translation system),又叫體外轉錄-翻譯的偶聯繫統,因為該系統需要製備無細胞提取物,還有人稱之為"溶胞粗製品翻譯系統"。
無細胞提取物的製備:
用機械的,超音波的,滲透壓或用適當的去污劑等方法,將細胞溶破,再高速離心出去其質膜與細胞核等.該提取液中含有RNA聚合酶,核糖體,tRNA和能量發生系統.
常見的體外無細胞翻譯系統:
兔網織紅細胞系統。網織紅細胞無細胞核,其合成的蛋白質90%以上為珠蛋白.
缺 點:已含有珠蛋白mRNA.可以在該體系中加入微球菌核酸酶和Ca2+,可很快分解,除去體系中的珠蛋白mRNA 。
麥胚系統用組織搗碎機將麥子搗碎,分離出麥胚.然後將粗製的麥胚加10倍體積的緩衝液與砂子共研磨.23000g離心所得的上清夜稱為S23 ;將S23分裝,保存於-20°C冰櫃中.
利用麥胚系統進行蛋白質翻譯合成研究時,需加的物質與網織紅細胞系統相似.由於該體系無mRNA,所以必須加入mRNA。
優 點:適於研究mRNA,活力強,價格低廉等.
缺 點:不同製品的活性差別較大,且合成分子量較大(71X105 Dal)的蛋白質時往往提前終止.
哺乳動物mRNA在紅細胞裂解液中翻譯
*SDS-PAGEanalysis of proteins translated by cell-free translation system. Lane 1: protein marker; Lane 2,without mRNA; Lane 3 to 7: translation products of total mRNA from mammalian cells
2、mRNA在無細胞體系中指導合成目的多肽的能力
3、mRNA分子的大小
哺乳動物mRNA長度為500-8000bp,大部分mRNA位於1.5-2.0kb之間.
4、總mRNA指導合成cDNA第一鏈長分子的能力
利用哺乳動物細胞提取的poly(A)+RNA合成cDNA
*Lane 1: -HindIII-EcoRI; Lane 2: the first chain of cDNA;
Lane 3: the second chain of cDNA; Lane 4: -HindIII
豐度
1)高豐度mRNA
目的mRNA在細胞中的含量占細胞質總mRNA量的50-90%, 該類mRNA在合成和克隆cDNA之前不需進一步純化特定mRNA .
2) 低豐度mRNA
目的mRNA在細胞中的含量占細胞質總mRNA量的0.5%以下.
5,mRNA的富集方法
典型的哺乳動物細胞含有10,000-30,000種不同的mRNA分子,某些mRNA分子在細胞內的拷貝數很低甚至只有一個拷貝.
mRNA的豐度與文庫克隆子數的關係
ln(1-P)
N= ln(1-1/n)
N: 所需克隆數; P: 要求的機率; n:一種mRNA在總mRNA中的相對比例
1) 按大小對mRNA進行分級分離
* 通過瓊脂糖凝膠電泳分離大小不同的mRNA分子,該方法的分離效果最好,但從凝膠中回收的得率較低.
* 蔗糖梯度離心:加入破壞RNA二級結構的變性劑如氫氧化甲基汞等,再進行蔗糖梯度離心以分離不同分子量的mRNA.
2) cDNA的分級分離
* mRNA通過反轉錄形成cDNA,在插入到克隆載體前,通過瓊脂糖凝膠電泳,將不同大小的cDNA分子分離開來.
* 優 點:
a)避免了分離過程中mRNA被污染的RNA酶降解
b)增加了獲得全長cDNA克隆的機率
c)獲得更準確的分級分離效果(分子量)
3)多聚核糖體免疫學純化法
* 使用抗體來純化合成目的多肽的多聚核糖體.將正在合成的新生多肽鏈的多聚核糖體結合到免疫親和柱(A蛋白-Sepharose柱)上,隨後用EDTA將多聚核糖體解離下來,並通過Oligo(dT)層析分離mRNA, 利用該方法可將目的mRNA純化數千倍. 目的mRNA在細胞中的含量可為數十拷貝.
分離
1.Oligotex mRNA Kits (QIAGEN)法
準備工作:
1.將Oligotex Suspension 置於37℃水浴中,旋轉混勻,溶解Oligotex.,然後置於室溫。
2.將OBB Buffer置於37℃水浴中,旋轉混勻,重溶沉澱物,然後置於室溫。
3.將OEB Buffer 置於70℃水浴中,待用。
試驗步驟:
表3:加入試劑量據此表
| Total RNA | RNase-free Water to: | Buffer OBB (ul) | Oligotex Suspension (ul) | Prep size |
| <=0.25mg | 250ul | 250ul | 15 | Mini |
| 0.25-0.5mg | 500ul | 500ul | 30 | Midi |
| 0.5-0.75mg | 500ul | 500ul | 45 | Midi |
| 0.75-1.00mg | 500ul | 500ul | 55 | Midi |
1. Total RNA 量不要多於1mg,用移液器取出所需RNA量到1.5ml離心管中,加RNase-free water 補足到500ul
2. 根據表3加入適當體積的OBB Buffer和Oligotex Suspension, 輕彈1.5ml離心管徹底混勻
3. 置於70℃水浴中3min
4. 取出置於室溫(20℃-30℃)10min
5. 13000rpm室溫離心 2min,用移液器吸出上清到一個新的1.5ml離心管中,保留上清直到polyA被結合上。
6. 用移液器取400ul OW2 Buffer混勻沉澱物,將混合物轉移到 Spin Column中,RT ,13000rpm,離心1min
7. 將Spin Column 轉移到一個新的1.5ml離心管中,加400ul OW2,RT, 13000rpm,離心1min
9. 取出25ul OER Buffer(70℃)到Column中,用移液器吹打3-4次樹脂,室溫 13000rpm,離心1min。
10. 測OD,並電泳定量。
2.磁珠法分離mRN
1. 在RNase-free的Eppendorf 管中加入0.1~1.0mg的總RNA和RNase-free水至終體積為500ul.
2. 65ºC加熱10分鐘。
3. 加入3ul生物素標記的Oligo(dT)和 13ul20×SSC於RNA中,輕輕混合,室溫放置逐漸冷去至室溫,一般需10 分鐘左右。
4. 同時配0.5×SSC 1.2ml和0.1×SSC 1.4ml.
5. 將磁珠(SA-PMPs)輕晃散開,放入磁性分離架上,使SA-PMPs集中於管的一側(約30sec),小心去上清,切不可離心。用0.3ml 0.5×ssc漂洗SA-PMPs,用磁性分離架集中磁珠,去除上清,重複3次。
6. 將漂洗後的SA-PMPs重新懸浮於0.1ml 0.5Xssc,注意漂洗後的SA-PMPs應在30分鐘內使用。
7. 將(3)中的oligo(dT)/mRNA退火反應物全部加入含漂洗好的SA—PMPs管中,輕輕搖勻,室溫下放10 分鐘。
8. 用磁性分離架捕獲SA-PMPs,小心去上清,但不要棄去。
9. 用0.1×SSC,每次0.3ml洗3次,每次都晃至SA-PMPs懸浮,最後一次漂洗後儘可能多的吸取水相,而不損壞SA-PMPs.
10.將SA-PMPs重新懸浮在0.1ml RNase-free水中,反覆顛倒,使SA-PMPs散開,洗脫mRNA。
11.用磁性分離架捕獲SA-PMPs,將洗脫的mRNA吸入另一個新的Eppendorf管中。
12.將SA-PMPs再懸浮於0.15ml RNase-free的水中,洗脫,與(11)步洗脫液合併。
13.將得到的mRNA溶液取幾微升跑電泳,若mRNA濃度不足以進行下一步的反轉錄,則需將得到的mRNA溶液濃縮(濃縮步驟見14-18)。
14.加0.1體積的3mol/l NaAc和1.0體積的異丙醇於洗脫液中,-20ºC沉澱過夜。
15.4ºC,13000g離心60 分鐘。去上清,加入500ul 70%乙醇混勻。
16.4ºC,7500g離心10 分鐘。
17.去上清,真空或空氣中自然風乾,但不要太乾,加適量RNase-free水溶解。
18.重複步驟5-12,將步驟8保留下的樣品重新上柱
注意事項:
1.所有操作均需要嚴格戴手套,戴口罩進行
2.如果total RNA質量高,雜質少,就選擇Oligotex的方法分離mRNA,相反則可以用磁珠法。
3.mRNA的電泳圖是smear。
TRNA
TRNA提取
試劑配製
準備工作:
1、研缽、5ml/10ml/ 25ml移液管、100ml/250ml量筒、250ml/100ml容量瓶、藥匙、試劑瓶等玻璃製品均用錫紙包裹口部,置於烤箱內,180℃,烤6小時。
2、50ml/1.5ml離心管、槍頭等塑膠製品用0.1‰DEPC水浸泡過夜後,121℃ 20mins 高壓滅菌。
3、電泳槽及電泳托、梳子用3%雙氧水處理。
4、常用試劑及其配方:
▲DEPC水:在1000ml去離子水中加入100ul DEPC, 靜置過夜後高壓滅菌。
▲0.78M檸檬酸納:PH=4~5
三水合檸檬酸納 22.94g
加DEPC水定容至100ml,室溫放置備用。
▲10%肌氨酸鈉:
肌氨酸鈉10g
加DEPC水定容至100ml,室溫放置備用。
▲變性裂解液:
0.78M檸檬酸鈉 8.25ml
10%肌氨酸鈉12.375ml
異硫氰酸胍118.05g
加DEPC水定容至 250ml,室溫放置備用
臨用前加β-巰基乙醇使其終濃度為1%(v/v)
▲ 2M 醋酸鈉 PH=4.5
NaAc·3H2O 13.6g
加DEPC水定容至50ml,高壓滅菌,室溫放置備用
▲3M醋酸鈉 PH=5
NaAc·3H2O 20.4g
加DEPC水定容至50ml,高壓滅菌,室溫放置備用
▲ 4M LiCL:
LiCL 24.164g
加DEPC水定容至100ml,高壓滅菌,室溫放置備用
▲0.5M EDTA PH=8.0
EDTA 18.61g
用NaOH調PH值至8.0,定容到100ml,高壓滅菌,室溫放置備用
▲10X MOPS (3-(N-嗎啉代)丙磺酸):
MOPS 41.86g
NaAC·3H2O 4.10g
0.5MEDTA(PH 8.0) 20ml
用NaOH調PH值 6.5 , DEPC水定容到1L,室溫避光放置備用。
▲ 1x MOPS:
10x MOPS 30ml
加DEPC水270ml,用時現配。
▲4x RNA Loading buffer:
10x MOPS 400ul
甘油(高壓過) 200UL
溴酚蘭 10ul
甲醛(37%) 72ul
去離子甲醯氨310ul
EDTA(0.5M PH 8.0) 8ul
ErBr(10mg/ml in DEPCH2O) 70ul
在4℃ 可保持3個月
▲ 10x PBS
pH=7.4
NaCl 80g
KCl 2g
Na2HPO4 14.4g
KH2PO4 2.4g
定容至 1000ml
▲變性電泳膠:
稱取0.5g瓊脂糖,加入47.5ml 1x MOPs,加熱至瓊脂糖熔化後,冷卻至50℃左右,加入2.5ml甲醛,輕輕混勻後倒入電泳托上。
▲變性電泳緩衝液:
在250ml容量瓶內加入5ml甲醛,用1x MOPS定容至250ml
2.動物組織t RNA的提取
1. 根據表1選擇適當的組織量和相應的變性裂解液量,將變性裂解液分裝到RNase-free的50ml無菌離心管中,冰浴5分鐘。
2. 將組織樣品放入變性裂解液中,在高速下勻漿15-30秒/次,直到看不見組織和細胞碎片。
3. 根據表1加入適量2M的乙酸鈉(pH4.0),反覆顛倒混勻4-5次。
4. 根據表1加入適量酚/氯仿,加蓋顛倒混合4-5次,再搖動10秒鐘。
5. 冰浴10分鐘。
6. 4°C,12000g離心20分鐘。
7. 小心轉移上層水相於另一個RNase-free的無菌離心管中,內含所需的RNA。蛋白質和DNA分別留在了有機相和中間層。
8. 加入等體積的異丙醇,-20°C沉澱30分鐘以上。
9. 4°C,12000g離心20分鐘。
10. 根據表1加入適量的變性裂解液重新溶解RNA。
11. 加等體積的氯仿,加蓋顛倒混合4-5次,再搖動10秒鐘。
12. 4°C,12000g離心20分鐘。
13. 小心轉移上層水相於另一個RNase-free的無菌離心管中,加入等體積的異丙醇,-20°C沉澱至少30分鐘。
14. 4°C,12000g離心20分鐘。
15. 棄上清,加1ml75%的乙醇漂洗RNA沉澱。4°C,12000g離心10分鐘。
16. 棄上清,空氣中乾燥RNA沉澱,直至沒有乙醇氣味。用適量DEPC水充分溶解RNA沉澱。
17. 取少量RNA用於測定OD值及電泳,其餘置-80°C冰櫃中保存。
表 1
| Mg# of tissue | 500 | 800 | 1000 |
| 變性裂解液(ml) | 5 | 8 | 10 |
| 2M NaOAC pH 4 (ml) | 0.5 | 0.8 | 1 |
| 水飽和酚 (mL) | 5 | 8 | 10 |
| 氯仿 (mL) | 1 | 1.6 | 2 |
| 異丙醇(mL) | 4 | 6 | 8 |
| 變性裂解液 (mL) | 0.5 | 0.8 | 1 |
| 異丙醇(mL) | 0.5 | 0.8 | 1 |
| 75% 乙醇(mL) | 1 | 1 | 1 |
| DEPC-treated H20 (mL) | 0.5 | 0.8 | 1 |
3.植物組織t RNA的提取
1. 先將研缽於-80℃冰櫃中預冷,然後將1g樣品在液氮中研磨成粉末狀。
2. 將粉末倒入盛有3ml變性裂解液的50ml離心管中,充分勻漿。(1g樣品加入3ml變性裂解液)。
3. 加入0.3ml(1/10體積)2M的NaAc(ph4-5)顛倒混勻。
4. 加入3ml(等體積)的水飽和酚,充分振盪,再加入1ml(1/3)的氯仿,振盪。
5. 冰浴10min。4℃,12000g離心10min
6. 小心轉移上清於另一離心管中,加入2倍體積的無水乙醇,-70℃沉澱至少1小時。
7. 4℃,12000g離心20min。
8. 去上清,取沉澱。加1ml 4M的LiCl 溶解沉澱(1mlLiCl/g組織),並轉入1.5ml離心管中。
9. 4℃,13000rpm離心15min。
10. 用0.4ml DEPC水溶解沉澱,加1/2體積的水飽和酚,1/2體積的氯仿,顛倒混勻。
11. 4℃,13000rpm離心10min。
12. 取上清,加1/10體積的3MNaAc(ph5)和2倍體積的無水乙醇,-70℃沉澱30min以上。
13. 4℃,13000rpm離心15min。
14. 棄上清,用1ml 75%的乙醇洗滌沉澱,4℃,13000rpm離心10min。
15. 棄上清,取沉澱,空氣中乾燥RNA沉澱,直至無乙醇味。
16. 用40-60ul DEPC水溶解(300-500ug/g)RNA沉澱。
17. 取少量RNA用於測定OD值及電泳,其餘置-80℃冰櫃中保存。
4. TRIzol Reagent 提取total RNA(GIBCO)
1. 查表2根據樣品量選擇適當的 TRIzol Reagent體積裝入50ml RNase-free離心管中,注意樣品體積不能超過TRIzol Reagent體積的10%。
2. 將組織樣品放入TRIzol Reagent中,高速下勻漿15-30秒/次,直至看不見組織和細胞碎片。
3.室溫溫育10分鐘 。
4. 據表2加入適量體積的氯仿,劇烈震盪15秒鐘,然後室溫下溫育3分鐘。
5. 4ºC,12000g離心15分鐘,形成淡紅色的苯酚/氯仿有機相,中間相和上層水相,水相約占TRIzol體積的60%。
6. 轉移上清於另一個Rnase-free的 50ml離心管中。根據表2加入適量異丙醇,-20ºC沉澱1小時以上。
7. 4ºC,12000g離心10分鐘。
8. 去上清,據表2加入適量體積的75%的乙醇混勻。
9. 4ºC,7500g離心5分鐘。
10. 去上清,空氣中乾燥或真空抽乾RNA沉澱,但不要太乾。加入適量體積的DEPC-WATER,溶解沉澱。
11. 取少量RNA用於測定其OD值和電泳,其餘置-80ºC冰櫃中保存。
表2
| 組織量 | TRIzol Reagent | 氯仿 | 異丙醇 | 75%乙醇 |
| 50~100mg | 1ml | 0.2ml | 1ml | 1ml |
cDNA
cDNA雙鏈合成
1. Superscipt II—RT合成第一鏈:
1. 在一RNase-free的0.2ml PCR管中,加入
xul mRNA(大約500ng)
1ul Xho I Primer(1.4ug/ul)
(5’ GAGAGAGAGAGAGAGAGAGAACTAGT CTCGAGTTTTTTTTTTTTTTTTTT…3’)
11-x ul RNase-free water
(大於500ng mRNA 分n管(500ng/tube)合成第一鏈, 第一鏈合成完畢後將n管合成一管進行第二鏈合成.)
2. 混勻後,70℃反應10分鐘;
3. 反應完成後,立刻將反應體系置於冰上5min;
4. 稍微離心一下,順序加入以下試劑:
4ul 5×first strand buffer
2ul 0.1M DTT
1ul 10mM dNTP(自己配製)
5. 混勻,稍微離心反應物之後,42℃放置2分鐘;
6. 反應完成,趁熱加入1 ul Superscipt II—RT,混勻;
7. 42℃反應50分鐘,然後70℃,15分鐘滅活反轉錄酶.
2. cDNA第二鏈的合成
1. 第一鏈反應完成後,取2ul一鏈產物-20℃冰櫃中保存,待電泳檢測。其餘的產物合併,混勻,然後順序加入下列試劑(promega):
20ul 10×DNA Polymerase I buffer
6ul 10mM dNTP(自己配製)
xul dd H2O
1ul RNase H(2U/ul)
10ul DNA Polymerase I(10U/ul)
總體系為200ul;
2. 混勻後,16℃反應2.5小時;
3. 70℃滅活10分鐘;
4. 反應完成後,得到200ul cDNA第二鏈反應體系,將此體系置於冰上;
5.取2ul二鏈產物,同保存的一鏈產物一起電泳鑑定。同時上1kb ladder,確定雙鏈的大小範圍。
註:一鏈,二鏈的電泳圖是smear,且二鏈稍比一鏈大一些。
3. 雙鏈cDNA末端補平
1. 在第二鏈反應體系中,順序加入下列試劑(promega):
6ul 10mM dNTP
2ul T4 DNA Polymerase(8.7U/ul)
2ul BSA(10mg/ml)
2. 稍微離心混勻反應物, 37℃反應至少30分鐘,然後75℃滅活10分鐘;
3. 加入等體積酚/氯仿/異戊醇,劇烈振盪後,常溫下13000g離心5分鐘;
4. 離心後,吸取上清於另一1.5ml eppendof管中,加入等體積氯仿,上下顛倒幾次混勻後,常溫下13000g離心5分鐘;
5. 吸取上清至另一eppendof管,加入1/10V3M NaAc(PH5.2)和2.5V預冷的無水乙醇,混勻,-20℃放置過夜以沉澱雙鏈cDNA;
6. 第二日,將昨日沉澱物在4℃,13000g離心60分鐘以充分沉澱雙鏈cDNA;
7.離心完畢,棄上清,加入1ml 70%乙醇洗滌沉澱,常溫下13000g離心5分鐘;
8.離心完畢,棄上清,乾燥沉澱至無乙醇氣味.
註:第3,第4步可以用PCR 純化試劑盒代替。
PCR純化試劑盒操作流程:
1.溶液PE使用前應加入適量體積95%-100%的乙醇,混勻。
2.向200ul二鏈補平產物中加入5倍體積的buffer PB,混勻。
3.加入spin column中,13000rpm離心1min。
4.加入0.75ml buffer PE,13000rpm離心1min。
5.13000rpm,再離心1min。
6.將spin column放入一新的離心管中,加入50ul buffer EB,靜置10min。
7.13000rpm離心2min。
8.加入30ul buffer EB,靜置10min。
9.13000rpm離心2min。
10.加入1/10體積3M的NaAc,2.5倍體積無水乙醇,混勻,-20℃沉澱過夜。
4 EcoR I adaptor 加接
1. 往雙鏈cDNA沉澱中加入9ul EcoR I adaptor(400ng/ul),4℃至少放置30分鐘以充分溶解cDNA沉澱;
2. 溶解完成後,順序加入下列試劑:
1.2ul 10×Ligase Buffer
1ul 10mM rATP
1 ul T4 DNA Ligase(4U/ul)
3. 混勻後,4℃連線3days,或者8℃過夜連線;
5 雙鏈cDNA末端的磷酸化及Xho I酶切
1. 連線反應完成後,將反應體系70℃放置15分鐘滅活T4 DNA Ligase;
2. 稍微離心使反應物集中至管底,室溫下放置5分鐘,然後加入下列試劑:
1ul 10×Ligase Buffer
1ul 10mM rATP
6ul dd H2O
1ul T4 PNK(10U/ul)
3. 37℃反應30分鐘,然後70℃滅活15分鐘;
4. 稍微離心使反應物集中至管底;
5. 室溫放置5分鐘;然後加入下列試劑:
4ul Xho 10×Buffer
2ul BSA
5ul ddH2O
8ul Xho I (10U/ul)
6. 37℃反應1.5小時,然後65℃滅活酶10分鐘;
7. 反應完成,雙鏈cDNA合成完畢。置於4℃準備回收。
6.膠回收cDNA
1.配製小膠數板(每個樣品一板):1%瓊脂糖凝膠,2ul EB/300ml 膠
2.取4℃保存樣品上樣,40ul/孔。
3.電泳50V;1hr
4.紫外燈下分別切下500~1kb、1.0-2.0kb及2.0-4.0kb cDNA 片段.,分別放入已做標記的1.5ml離心管中。
5.稱取膠重,加入三倍體積buffer QXI(例如,100mg膠中加入300ul buffer QXI)
6.50℃ 水浴數分鐘,至膠完全融化。用手指彈 QIAEX II 使重懸,每管中加入5ul QIAEXII
7.50℃ 水浴10min,每隔2min 取出顛倒混勻數次,使QIAEX II 保持懸浮
8.4℃,13000rpm,30sec。(棄上清,離心機中甩一下,吸取上清)
9.加入500ul buffer QXI,輕彈管底使QIAEX II 重懸
10. 離心並去上清(同操作8)
11. 加入500ul buffer PE,重懸QIAEX II,離心30sec,去上清
12. 再加入500ul buffer PE,重懸QIAEX II,離心30sec,棄上清,離心機中甩一下,吸去上清
13. 超淨台上吹乾(至無乙醇味),加入10ul elution buffer,重懸QIAEX II,靜置5min,13000rpm,30sec。吸上清,冰上放置。
14. 取1ul上清上樣電泳,同時做分子量標準(1kb ladder)及DNA含量標準(10ng,20ng)作對照。
15. 將收回的cDNA置於-20℃內保存,據電泳結果,取適量DNA進行連線。
注意事項:
1.膠回收前電泳槽,電泳板,梳子等都要用1%的HCl浸泡過夜。
2.膠回收時電壓要穩定。
第一鏈
第一鏈的合成
oligo(dT)引導的DNA合成法:
利用真核mRNA分子所具有的poly(A)尾巴的特性,加入12-20個脫氧胸腺嘧啶核苷組成的oligo(dT)短片段,由反轉錄酶合成cDNA的第一鏈.
缺 陷:
因為逆轉錄酶無法到達mRNA分子的5'-末端,必須從3'-末端開始合成cDNA.對於大分子量的較長的mRNA分子而言,特別麻煩.
隨機引物引導的cDNA合成法 (randomly primed cDNA synthesis) :
根據許多可能的序列,合成出6-10個核苷酸長的寡核苷酸短片段(混合物),作為合成第一鏈cDNA的引物.在套用這種混合引物的情況下,cDNA的合成可以從mRNA模板的許多位點同時發生,而不僅僅從3'-末端的oligo(dT)引物一處開始.
第二鏈
第二鏈的合成
第一鏈的合成反應完成後,得到DNA/RNA雜交分子,在DNA聚合酶的作用下,以第一鏈為模版,合成cDNA的第二鏈。
自身引導
變性降解除去雜交分子中的RNA,單鏈cDNA的3'端能夠形成髮夾狀的結構作為引物,在大腸桿菌聚合酶I Klenow或反轉錄酶的作用下,合成cDNA的第二鏈.
缺 點:
在以S1核酸酶切割cDNA的髮夾狀結構時,會導致對應於mRNA 5'端的地方的序列出現缺失和重排. S1核酸酶的純度不夠時,會偶爾破壞合成的雙鏈cDNA分子.
自身引導法合成雙鏈cDNA
置換合成
原 理:
以第一鏈合成產物cDNA:mRNA雜交體作為切口平移的模板,RNA酶H在雜交體的mRNA鏈上造成切口和缺口,產生一系列RNA引物,在大腸桿菌DNA聚合酶I的作用下合成cDNA的第二鏈.
優點:a)合成cDNA的效率高
b)直接利用第一鏈的反應產物,不需純化
c)避免使用S1核酸酶來切割雙鏈cDNA
3,引物-銜接頭法
克隆
將合成的雙鏈重組到質粒載體或噬菌體載體上,轉化大腸桿菌寄主細胞增殖.
1,同聚物加尾法
利用小牛胸腺末端轉移酶在雙鏈cDNA和質粒載體的3'-端都加上一個互補的同聚片段,通過退火使兩個片段連線成重組質粒,再將重組質粒轉化受體細胞.
同聚物加尾法克隆雙鏈cDNA
2,接頭-銜接頭法
3,mRNA-cDNA克隆法
方 法:
在mRNA反轉錄合成cDNA第一鏈後,將dA殘基加到mRNA:cDNA雜交體上,然後與帶dT尾的克隆載體連線,轉化受體細胞,在宿主細胞內,mRNA被降解並代之以DNA.
缺 點:
克隆的效率低(為雙鏈cDNA克隆的1/10)
4,Okayama-Berg法合成並克隆雙鏈cDNA
篩選鑑定
1 核酸雜交
是最常用,最可靠的方法之一.可大規模地分析文庫的克隆子.
1)同源探針:至少含有所需cDNA克隆的一部分確切序列.常用於以一個部分克隆分離cDNA文庫中的全長克隆.
2)部分同源探針:探針的序列與所要篩選的cDNA克隆的序列相關但不相同.常用於克隆家族基因.
3)總cDNA探針:
通過反轉錄酶均勻摻入放射性核苷酸或通過總的或分級分離得到的poly(A)+mRNA進行末端標記而獲得cDNA探針.
cDNA扣除探針:
從第一種mRNA製備cDNA探 針, 連續多次與20倍過量的第二種mRNA雜交;
回收未雜交的cDNA探針, 再與100倍過量的第一種mRNA雜交, 使得原mRNA中的一些特異序列得到高度富集.
* 主要用於探測cDNA文庫中與調節水平有所差別的mRNA克隆.
5)合成寡核苷酸探針
2 特異性免疫學檢測
在cDNA表達文庫中,目的基因的表達產 物能與特異性抗體發生免疫學反應,通過酶學的方法加以檢測
3 cDNA克隆的同胞檢測
將cDNA文庫分成若干組含有10-100個克隆子的易於處理的亞cDNA文庫,對每組亞cDNA文庫進行檢測,當鑑定出陽性庫後再不斷將其分成更細的庫進行檢測,直到獲得陽性單克隆.
4 cDNA克隆的確證
cDNA克隆含有編碼某一特定蛋白質的完整胺基酸序列的開放讀框.
第四節 目的基因的分離
外源基因:
插入到載體內的那個特定的片段基因.
目的基因:
那些已被或者準備要分離,改造,擴增或表達的特定基因或DNA片段.
載體製備
1.pBlueScriptII的提取
1.取1ul商品的pBlueScriptII,轉化入大腸桿菌宿主菌中,取5ul轉化產物均勻塗布在含AMP的LB平板上,37℃培養過夜。
2.第二天取一隻無菌的50ml離心管,加入10ml AMP抗性的LB液體培養基,挑單克隆於離心管中,37℃,250rpm,培養過夜。
3.第三天取200µl小搖後的菌液接種於250ml 含AMP的LB液體培養基中,37℃,250rpm培養6 hr左右,使OD值達到0.6-0.8。
4.將菌液移入250ml離心管中, 4℃,3000rpm,離心15min。取出離心管,菌團朝上倒掉上清,將離心管倒置於吸水紙上使上清充分濾乾。(注意離心前需配平)
5.加入10ml溶液Ⅰ(50mM Glucose , 25mM Tris-HCl,10mM EDTA, pH8.0),加入RNase至終濃度100µg/ml,晃動搖菌,使菌體充分懸浮,靜置10min。
6.按NaOH(0.4N):SDS(2%)--1:1的比例新鮮配製溶液Ⅱ,加入20ml溶液Ⅱ,靜置3-5min。
註: 靜置時間勿超過5min,提前將溶液Ⅲ置於冰盒中。
7.加入15ml冰浴的溶液Ⅲ,冰浴15-30min。
8.4℃,5000rpm,離心15min。
9.取上清於兩個50ml離心管中,棄去原離心管中的沉澱。
10.每管加入0.6倍體積的異丙醇,充分混勻,室溫下放置10min。
11. 20℃,12000g,離心20min回收質粒沉澱。
12.棄上清,用70%的乙醇洗2次。
13.棄上清,倒扣於吸水紙上,儘量空乾液體。
14.用3ml TE(pH8.0)溶解沉澱,移入1.5ml Eppendorf離心管中。
15.電泳檢查DNA質量並定量。(必要的話,可以用膠回收的方法先純化一下質粒再進行雙酶切。)
2.pBlueScriptII的雙酶切消化
1.以如下體系進行EcoRI酶切:
pBSK(+) X µl(6µg)
ddH2O 174-X µl
10×Buffer E 20 µl
混勻,加入限制性內切酶:
EcoRI (10U/ µl) 6 µl
總體積為200 µl。
2.輕彈管壁或用槍頭輕輕吹打混勻,在離心機上甩一下。
3.37℃,水浴1hr。
4.加入200ul 1:1的酚/氯仿,混勻。4℃,13000rpm,離心15min。
5.取上清,加入等體積的氯仿,4℃,13000rpm,離心10min。
6.取上清,加入0.1倍體積的NaAC和2.5倍體積的無水乙醇,-20℃,沉澱30min。
7. 4℃,13000rpm,離心10min,棄上清,取沉澱。
8.加入200ul 70%的乙醇洗沉澱。
9.4℃,13000rpm,離心10min,棄上清,取沉澱。
10.自然風乾沉澱,至無乙醇味,加入100ul ddH2O充分溶解沉澱。
11.加入以下試劑進行XhoI酶切:
ddH2O 74 µl
10×Buffer D 20 µl
混勻,加入限制性內切酶XhoI:
XhoI (10U/ µl) 6 µl
總體積為200 µl。
12.輕彈管壁或用槍頭輕輕吹打混勻,在離心機上甩一下
13.37℃,水浴1.5hr。
14.加入200ul 1:1的酚/氯仿,混勻。
15.4℃,13000rpm,離心15min。
16.取上清,加入等體積的氯仿,4℃,13000rpm,離心10min。
17.取上清,加入0.1倍體積的NaAC和2.5倍體積的無水乙醇,-20℃,沉澱30min。
18. 4℃,13000rpm,離心10min,棄上清,取沉澱。
19.加入200ul 70%的乙醇洗沉澱。
20.4℃,13000rpm,離心10min,棄上清,取沉澱。
21.自然風乾沉澱至無乙醇味,加入40ulddH2O充分溶解沉澱,得到雙酶切載體。
3.載體去磷酸化
1.在40ul雙酶切載體中加入以下試劑:
6ul 10×buffer
6ul CIAP(0.01U/ul)
8ul ddH2O
總體積60ul。
2.輕彈管壁或用槍頭輕輕吹打混勻,在離心機上甩一下。
3.37℃,水浴1hr。
4.70℃,15min,滅活酶。
5.電泳分離,膠回收雙酶切載體,定量。
4.載體效率檢測
1.按以下所示作4個連線反應:
| DNA | 連線酶 | ||
| 1 | pBlueScriptII/E/X /CIAP | - | 檢測酶切效率 |
| 2 | pBlueScriptII/E/X /CIAP | + | 檢測脫磷效率,載體自連效率 |
| 3 | 商品Vector加標準Insert | + | 對照 |
| 4 | 自製Vector加標準Insert | + | 檢測載體效率 |
14℃連線過夜。
2. 各取1ul連線產物作電轉化(具體流程見電轉化)
3.計算克隆數,計算載體相對脫磷效率及連線效率。
鳥槍法
又叫霰彈法,其特點是繞過直接分離基因這一關.由於目的基因在整個基因組中太少太小,在相當程度上靠"運氣".
原 理:
基因組DNA
物理(剪下力,超音波等) 或生化方法(限制性內切酶)切割
長度與一般基因大小相當的DNA片段的混合物
隨機地重組入適當的載體
轉 化
大腸桿菌中擴增
適當的方法篩選
要求有簡便的篩選方法:
利用特定基因缺陷型(如營養缺陷型等)的受體細胞,或特定的寡核苷酸DNA 片段探針以特定基因產物的抗體,可通過雙表型的篩選或用分子雜交技術,免疫篩選技術檢出目的基因.
示例:麵包酵母吲哚甘油磷酸脫氫酶基因的製取
EcoRI 載體
酵母DNA 片段基因 重組DNA
轉化
"吲哚甘油磷酸脫氫酶型組氨酸缺陷型"大腸桿菌
基本培養基
篩選,分離菌株
目的基因
物化方法
基因工程初始階段所用的方法,已不用.利用核酸雙螺旋之間存在著鹼基G C,A T配對特性,分離目的基因.
例如:海膽rDNA分子內其G C含量可以達63%(其穩定性高,溶解溫度高),通過熱變性和S1酶解處理可得到提純50倍的rDNA,最後經氯化銫平衡梯度離心,得到相對分子量為1.9X107Dal的高純rDNA.
從基因文庫中分離目的基因
分離基因
如果手中有足夠量可產生抗體的來源於真核細胞的蛋白質,可以通過雙抗體免疫法分離出此蛋白的基因.
基本原理:
核糖體沿mRNA進行多肽鏈合成時形成多聚核糖核蛋白體,而具有不同長度的新生肽鏈在核糖體上不斷延伸;
將從細胞勻漿液中製備出的多聚核糖核蛋白體同特定抗體一起保溫,形成多聚核糖體同抗體的複合體;
當加入特定蛋白的抗體產生的第二抗體時,產生沉澱,就可以通過不連續蔗糖梯度離心,將所要的含有特定的mRNA的多聚核糖體同總多聚核糖體分離;再通過酚,氯仿抽提去除蛋白及oligo-dT柱親合層析,就可以得到為特定蛋白編碼的mRNA,再通過反轉錄得到cDNA.
化學合成
基因的化學合成
基因片段的全化學合成
首先合成一個基因的所有片段,相鄰的片段間有4—6個鹼基的重疊互補,退火後,用T4DNA連線酶將各片段以磷酸二酯鍵的共價鍵形式連線成一個完整的基因.
基因的化學—酶促合成
不需要合成完整基因的所有寡核苷酸片段,而是合成其中一些片段,相鄰的3'-末端有一短的順序相互補,在適當的條件下通過退火形成模板—引物複合體,然後在存在四種的條件下,用大腸桿菌DNA聚合酶I大片段填補互補片段之間的缺口,最後用T4DNA連線酶連線及適當的限制性內切酶.
PCR
PCR技術的套用
目的基因的直接克隆
與常規的基因克隆方法相比,PCR的優點是快速,簡單,但是用PCR克隆目的基因的限制是必須知道側接靶序列的核苷酸序列,以製備引物.因而該法具有很大的局限性.
可直接利用具有平端的PCR產物進行克隆,但利用合適的引物,在待克隆的目的基因二側引入不同的限制性酶切點,則可將擴增之後的目的基因定向克隆到載體中,避免載體的自身環化,提高克隆效率.
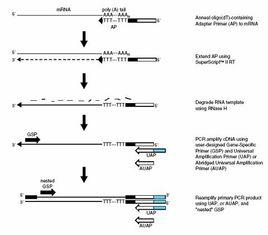
cDNA的克隆
利用PCR技術,只需增加一步逆轉錄反應,便可從少數mRNA的構建cDNA文庫,以mRNA為模板,以oligo(dT)為引物,在依賴於RNA 的DNA聚合酶催化下體外合成cDNA第一鏈之後,可通過PCR擴增此鏈.
如在此鏈的3'端再加上一段鳥苷酸殘基同聚物,則可以使用oligo(dT)和oligo(dC)作為後續PCR擴增的引物,也可酌情在這些引物的5'端加上限制性酶切點,以利於將所得的雙鏈DNA克隆到適當的載體中.
在某些情況下,如已知RNA(或其基因)兩端的核苷酸序列,mRNA5'端核苷酸序列或其編碼的蛋白質N端的胺基酸序列,便可設計特定的兩端引物,用於直接克隆特定的目的基因cDNA,從而省略從cDNA文庫中篩選cDNA克隆等序列費時的操作.
分子生物學實驗技術
| 分子生物學(molecularbiology) 是從分子水平研究作為生命活動主要物質基礎的生物大分子結構與功能,從而闡明生命現象本質的科學。而分子生物學的各種實驗方法,是本學科得以高速發展和不斷取得突破性成就的基礎。隨著學科的發展和各相關交叉學科的進步,分子生物學實驗技術必定會越來越高效與精準,成為人類探索自然、改善自然的有力工具。 |