概述
 蛋白質二級結構
蛋白質二級結構蛋白質二級結構
二級結構在生物化學及結構生物學中,是指一個生物大分子,如蛋白質及核酸(DNA或RNA),局部區段的三維通式。然而它並不描述任何特定的原子位置(在三級結構中描述)。二級結構是由生物大分子在原子解析度結構中所觀察到的氫鍵來定義的。蛋白質的二級結構通常是以主鏈中氨基之間的氫鍵模式來定義〈與主鏈-側鏈間以及側鏈-側鏈間的氫鍵無關〉,亦即DSSP的定義。[1]而核酸的二級結構是以鹼基之間的氫鍵來定義。
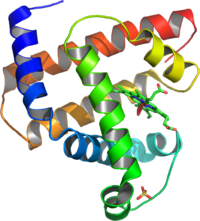 肌紅蛋白的立體圖
肌紅蛋白的立體圖對生物大分子的二級結構含量可以以光譜來初步估計。對於蛋白質,最常用的方法是圓二色譜(利用長紫外線,波長範圍170-250nm)。在獲得的光譜吸收曲線上,α螺鏇結構會在208nm及222nm兩處同時出現極小值,而204nm和207nm處出現單個極小值則分別表示存在無規捲曲和β摺疊結構。另一個較常用的方法是紅外光譜,它可以偵測因氫鍵所造成胺基的震盪。而光譜中,測定二級結構最準確的方法是利用核磁共振光譜所紀錄的化學位移,由於儀器和樣品製備上的原因,這一方法較為少用。
常見蛋白質二級結構
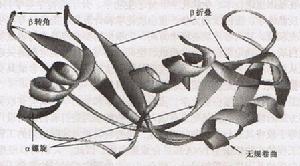
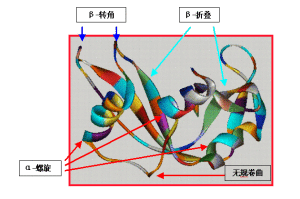
α-螺鏇(α-helix)蛋白質中常見的一種二級結構,肽鏈主鏈繞假想的中心軸盤繞成螺鏇狀,一般都是右手螺鏇結構,螺鏇是靠鏈內氫鍵維持的。每個胺基酸殘基(第n個)的羰基氧與多肽鏈C端方向的第4個殘基(第n+4個)的醯胺氮形成氫鍵。在典型的右手α-螺鏇結構中,螺距為0.54nm,每一圈含有3.6個胺基酸殘基,每個殘基沿著螺鏇的長軸上升0.15nm。螺鏇的半徑為0.23nm。
β-摺疊(β-sheet)
是蛋白質中的常見的二級結構,是由伸展的多肽鏈組成的。摺疊片的構象是通過一個肽鍵的羰基氧和位於同一個肽鏈或相鄰肽鏈的另一個醯胺氫之間形成的氫鍵維持的。氫鍵幾乎都垂直伸展的肽鏈,這些肽鏈可以是平行排列(走向都是由N到C方向);或者是反平行排列(肽鏈反向排列)。
β-轉角(β-turn)
多肽鏈中常見的二級結構,連線蛋白質分子中的二級結構(α-螺鏇和β-摺疊),使肽鏈走向改變的一種非重複多肽區,一般含有2~16個胺基酸殘基。含有5個胺基酸殘基以上的轉角又常稱之環(loops)。常見的轉角含有4個胺基酸殘基,有兩種類型。轉角I的特點是:第1個胺基酸殘基羰基氧與第4個殘基的醯胺氮之間形成氫鍵;轉角II的第3個殘基往往是甘氨酸。這兩種轉角中的第2個殘基大都是脯氨酸。
蛋白質
蛋白質是以胺基酸為基本單位構成的生物大分子。一級結構:蛋白質多肽鏈中胺基酸的排列順序,以及二硫鍵的位置。二級結構:蛋白質分子局區域內,多肽鏈沿一定方向盤繞和摺疊的方式。三級結構:蛋白質的二級結構基礎上藉助各種次級鍵捲曲摺疊成特定的球狀分子結構的空間構象。四級結構:多亞基蛋白質分子中各個具有三級結構的多肽鏈,以適當的方式聚合所形成的蛋白質的三維結構。用約20種胺基酸作原料,在細胞質中的核糖體上,將胺基酸分子互相連線成肽鏈。一個胺基酸分子的氨基,脫去一分子水而連線起來,這種結合方式叫做脫水縮合。通過縮合反應,在羧基和氨基之間形成的連線兩個胺基酸分子的那個鍵叫做肽鍵。由肽鍵連線形成的化合物稱為肽。蛋白質二級結構預測
早期蛋白質二級結構預測的方法是建基於胺基酸形成螺鏇或摺疊的傾向,而有時須聯同估計形成二級結構的能量的方法來使用。這些方法在預測殘基的三種狀態(螺鏇、摺疊或捲曲)可以有約60%的準確性,若使用多序列比對可以將準確性大幅提升至80%。多序列比對可以知道胺基酸在某一位置的完正分布(包括在其附近的位置,一般在每一邊的7個殘基),而演化過程提供了結構趨向更明確的圖畫。例如,在蛋白質某位置的甘胺酸,本身已表明那是一個任意形。但是多序列對比可以發現,在接近十億年演化後95%的蛋白質中,那是一個有利螺鏇的胺基酸。再者,若在那位置檢測平均疏水性,亦會發現其殘基可溶性是與α螺鏇一致。綜合來說,這些因素顯示原先蛋白質內甘胺酸是α螺鏇結構,而非任意形。多種方法都會結合已有的數據來組成三種狀態的預測,這些方法有神經網路、隱馬爾可夫模型及支持向量機。現代預測方法亦可在每一個位置的預測結果提供信賴分數。二級結構預測方法一直不斷地在校準,例如EVA實驗。基於約270個星期的測試,最準確的方法要算是PsiPRED、SAM、PORTER、PROF及SABLE。有趣的是,在這多種方法中找出共識或一致,並不能提升它們的準確性。最大改善的地方似乎是在β股的預測,因為所使用的方法會忽視一些β股段。整體上而言,最高的預測準確性只可以達90%,因DSSP的標準方法的性質,與校準的預測相違背。
準確的二級結構預測是三級結構預測的重要原素。例如一個確定的βαββαβ二級結構模式,就是鐵氧化還原蛋白的記號。
內部連結
蛋白質一級結構
在生物化學裡,生物分子的一級結構是其分子組成和分子間化學鍵結的精確模樣。對於一典型的無分支、無交叉的生物聚合物(如DNA、RNA或典型的細胞內蛋白質等分子),其第一結構等同於描述其單體單位的序列,即如DNA序列和肽序列。「一級結構」這一名詞在Linderstrom-Lang於1951年的LaneMedicalLectures上首次被提到。一級結構和一級序列有一點相似,即使在二級或三級結構中並沒有平行的概念。蛋白質三級結構
在生物化學里,蛋白質的三級結構是指其整體形狀,亦稱為其摺疊。蛋白質分子是一連串的胺基酸一條線地接結,基本上假定其會有一可作用其生物功能的三維結構。對蛋白質三級結構的研究稱為結構生物學。蛋白質四級級結構
四級結構是生物化學中用於描述多亞基蛋白質複合物中各個摺疊蛋白質亞基的排列組合。結構生物學
結構生物學是一門以生物物理學和生物化學手段來研究生物大分子(如蛋白質分子和核酸分子)的三維結構(包括構架和形態),並研究結構與對應功能的關係的學科。由於結構生物學能夠解釋生物大分子的構象和相互作用的方式,而所有的生命活動都是通過各種生物大分子的相互作用來實現;因此,對於生物學家們來說,這是一個非常有吸引力的領域。蛋白質立體結構原則
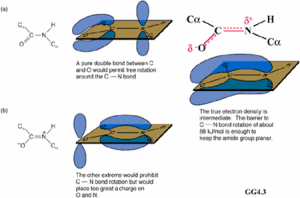 肽鍵
肽鍵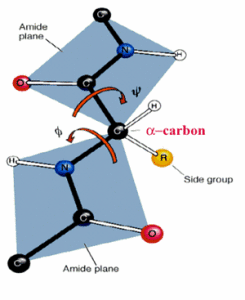 肽單元或肽鍵平面
肽單元或肽鍵平面