簡介
 RNA聚合酶
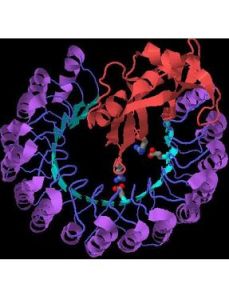
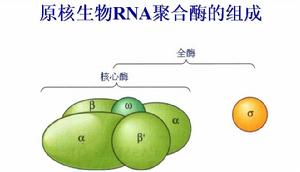
RNA聚合酶細菌的RNA聚合酶,像DNA聚合酶一樣,具有很複雜的結構。其活性形式(全酶)為15S,由5種不同的多肽鏈構成,按分子量大小排列分別為β'(155000),β(151000),σ(7000),α(36500)和ω(11000)。每分子RNA聚合酶除有兩個α亞基外,其餘亞基均只有一個,故全酶為β'βα2σω(450000)

如果全酶為球狀,則可計算其直徑約為100A,即約為雙鏈DNA的30bp節段的長度。然而全酶可結契約60個核苷酸,提示其形狀應為橢圓球形。
σ亞基和其他肽鏈的結合不很牢固。當σ亞基脫離全酶後,剩下的β'βα2ω稱為核心酶。核心酶本身就能催化核苷酸間磷酸二酯鍵的形成,不論有無σ存在的利福平(rifampicin)和利福黴素(rifamycin)能結合在β亞基上而對此酶發生強烈的抑制作用。它抑制RNA合成的起始而不抑制其延長。β亞基的基因rpoB的突變可使細菌對利福平有抗性。而另一種抗生素鏈霉溶菌素(streptolydigin)能抑制RNA鏈的延長,rpoB的突變卻可使細胞對鏈霉溶菌素亦發生抗性。總的看來β亞基似乎是酶和核苷酸底物結合的部位。肝素能與DNA競爭與RNA聚合酶相結合。肝素是一種多價陰子,能和β'亞基結合,從而在體外抑制轉錄作用。可見β'亞基的鹼性較強,適於與模板DNA相結合。α亞基的功能現尚未知。然而,當噬菌體T4感染E.coli時,其α亞基即在精氨酸上被ADP-核糖化。這種修飾作用使該RNA聚合酶全酶對其原先識別的啟動子的親和力減弱。所以有人認為,α亞基的功能可能是識別其相應的啟動子。
結構
 RNA聚合酶
RNA聚合酶相比之下,宿主細胞的RNA聚合酶卻能轉錄細胞內的許多轉錄單位(>1000個)中的任意一個。這些轉錄單位中有一些可由RNA聚合酶直接轉錄,不需要其他因子的幫助。但大多數這些轉錄單位僅在更多的蛋白質因子存在下才能被該RNA聚合酶所轉錄。所以,可以想像宿主細胞所以要求這樣大和複雜,至少部分反映了它需要和許多其他因子相互作用,而不是其催化活性的固有的要求。
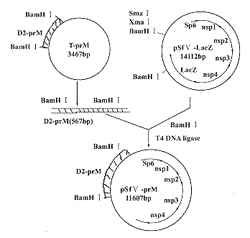
E.coli的RNA聚合酶各亞基的基因(除ω亞基的基因尚不詳外)均存在於三個操縱子中,這些操縱子還含有蛋白質合成和DNA合成所需的基因。P71主要啟動子(P)均在操縱子的左側,RNA則向右方合成。次要啟動子(p)包括σ操縱子中的一個啟動子(PHS,是在熱休克條件下才有活性的啟動子),均位於操縱子之內。這些次要啟動子僅啟動在其右側的基因。β操縱子的前面兩個基因L11和L1,有時被認為組成另一個獨立的操縱子,因為在L10基因之前另有一啟動子。這些基因編碼50個左右核糖體蛋白質中的某些蛋白質。σ操縱子中還含有複製所需的蛋白質,即DNA引物酶的基因。現在還不清楚這些操縱子是如何進行調節的。
 RNA聚合酶
RNA聚合酶啟動環功能
 RNA聚合酶
RNA聚合酶以往研究表明,TL結構直接與新合成RNA的3'端和NTP相互作用,其中一個保守的組氨酸殘基直接與NTP底物相結合。Kaplan等人用其他胺基酸替代釀酒酵母RNA聚合酶Ⅱ中TL結構中的His1085殘基,結果導致釀酒酵母表現出嚴重的生長缺陷,甚至無法存活。離體實驗表明,His1085能夠選擇正確的NTP底物,該位點變異後,聚合酶Ⅱ摻入錯誤的NTP底物和2'dNTP底物的可能性大大增加。
α鵝膏覃鹼能夠抑制RNA聚合酶Ⅱ的延伸速率,降低其底物選擇性,但His1085被替代後的RNA聚合酶Ⅱ對α鵝膏覃鹼則具有較高的抗性。研究人員進一步研究了純化的RNA聚合酶-α鵝膏覃鹼複合體的晶體結構,發現α鵝膏覃鹼和TL結構間存在直接的相互作用。因此,研究人員認為,α鵝膏覃鹼通過直接作用於TL結構而降低RNA聚合酶Ⅱ的速率和保真度。
代換
 RNA聚合酶
RNA聚合酶T7是E.coli的烈性噬菌體,基因組長為39,936nt,順序已知。整個基因組可能具有55個基因,其中44個基因的產物已鑑別出了,已知30種是有功能的轉錄可分為3組,30°C整個生活周期約為25¢分鐘,但其DNA的注入很慢,整個的過程約10分鐘,而其它的噬菌體僅1分鐘即可完成。這也是一種調控的手段,因未注入宿主細胞中的DNA是不能轉錄的。在感染6分鐘後,E.coli的RNA合成體系全部被T4的合成體系所取代。
在感染後的前6分鐘,T7噬菌體的RNAPol尚未表達,因此仍使用了宿主E.coli的RNA聚合酶,轉錄的基因主要有10個(0.3,0.4,0.5,0.6,0.65,0.7,1,1.1,1.2,1.3),其0.3基因編碼宿主限制酶的抑制蛋白,使T7進入宿主免遭降解;0.7基因編碼一種蛋白激酶,能將E.coli的RNA聚合酶磷酸化,為抑制宿主RNAPol作準備;基因1編碼T7RNAPol聚合酶,分子量為98KDa,是一條單鏈肽,它所識別的啟動子是由23bp組成的保守順序(-17~16)。
晚期轉錄分為二組(Ⅱ和Ⅲ)。感染後6~12分鐘後T7就利用自己的RNA聚合酶轉錄第Ⅱ組轉錄物。這一組約含(1.4,1.5,1.6,1.7,1.8,2,2.5,2.8,3,3.5,3.8,4,5,5.7,6),其中對轉錄調控有關係的是基因2,其產物是E.coli聚合酶抑制劑。宿主的RNA聚合酶先受到基因0.7產物的磷酸化,再經抑制完全失去了作用,此時T7已不再需要I組的轉錄物了,因此廢除了宿主RNA聚合酶的功能,而將自己編碼的RNA聚合酶取而代之。第Ⅱ組的基因多與T7的複製酶系的合成及幾種結構蛋白有關,但第Ⅱ組的啟動子和複製的φL啟動子因其保守順序中分別有2~7個鹼基發生了改變,所以是較弱的啟動子,但由於其位置排在Ⅲ組啟動子的前面,所以先進入宿主得以轉錄,而不與第Ⅲ組啟動子的競爭。
 RNA聚合酶
RNA聚合酶第Ⅰ組轉錄的終止位置在鄰近1.4基因的上游區(1.3和1.4之間),此是E.coliRNA聚合酶的終止子)它解離後T7RNA聚合酶則重新從第Ⅱ組起始轉錄。第Ⅱ組和第Ⅲ組都使用終止子Tj,由於自注入宿主緩慢,在第Ⅱ組轉錄時第Ⅲ組基因尚未注入,仍得不到表達。
Tj的終止效率是90%,尚有10%可以延長轉錄T7的末端,Tj位於基因10和11之間。基因10是主要的頭部蛋白,需要量大,它排在第Ⅱ組末是合理的,即有強啟動子的啟動,又在轉錄後被終止,這樣不僅滿足了裝配顆粒的需要,又不至造成浪費。而排在基因10後面的基因都是編碼少量頭部蛋白和尾部蛋白的,無需大量的轉錄,後面越過Tj延長轉錄的強度就可滿足需要了,這本身也是一種終止子對轉錄的調控。
作用
 RNA聚合酶
RNA聚合酶真核生物的RNA聚合酶分三類。RNA聚合酶Ⅰ存在於核仁中,轉錄rRNA順序。RNA聚合酶Ⅱ存在於核質中,轉錄大多數基因,需要“TATA”框。RNA聚合酶Ⅲ存在於核質中,轉錄很少幾種基因如tRNA基因如5SrRNA基因。有些重複順序如Alu順序可能也由這種酶轉錄。上面提到的“TATA”框又稱Goldberg –Hogness順序,是RNA聚合酶Ⅱ的接觸點,是這種酶的轉錄單位所特有的。它在真核生物的轉錄基因的5’端一側,在轉錄起點上游20至30個核苷酸之間有一段富含AT的順序。如以轉錄起始點為0,則在-33到27個核苷酸與-27至21核苷酸之間,有一個“TATA”框。一般是7個核苷酸。原核生物中也類似“TATA”框的結構。RNA聚合酶作用在“TATAAT”(Pribnow)盒和“TTGA-CA”框附近。
研究進展
 RNA聚合酶
RNA聚合酶RNA聚合酶(RNAP),只要是稍微接觸過分子生物學的人就不會對這個名詞陌生,在DNA要將它的遺傳信息表達出去,成為真正具有功能效應的單位——蛋白的時候,需要中間體RNA的幫助,而要實現這個過程就不能缺少這個重要的酶。StevenBlock和他的同事利用一種比之前光阱(opticaltrap)更穩定的裝置發現在大腸桿菌轉錄的過程中,這個酶每隔大約1000個鹼基就會出現一個錯誤,但是RNAP也有良好的自我糾正機制,能及時發現和糾正這些錯誤。
 RNA聚合酶
RNA聚合酶這一研究獲得了許多科學家的認可,德克薩斯大學健康中心的RuiSousa認為這是一項迄今為止最好的布朗荊輪的證明,但是也有科學家對此表示懷疑態度。