簡介
 基因晶片技術
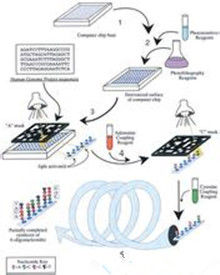
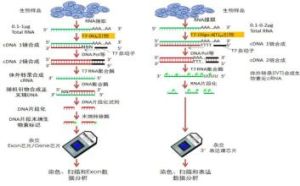
基因晶片技術基本原理
 基因晶片技術
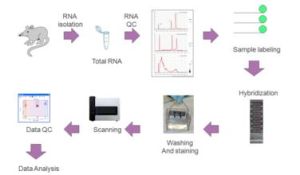
基因晶片技術晶片基片可用材料有玻片、矽片、瓷片、聚丙烯膜、硝酸纖維素膜和尼龍膜,其中以玻片最為常用。為保證探針穩定固定於載體表面,需要對載體表面進行多聚賴氨酸修飾、醛基修飾、氨基修飾、巰基修飾、瓊脂糖包被或丙烯醯胺矽烷化,使載體形成具有生物特異性的親和表面。最後將製備好的探針固定到活化基片上,目前有兩種方法:原位合成和合成後微點樣。根據晶片所使用的標記物不同,相應信號檢測方法有放射性核素法、生物素法和螢光染料法,在以玻片為載體的晶片上目前普遍採用螢光法。相應螢光檢測裝置有雷射共聚焦顯微鏡、電荷偶合器(chargecoupleddevices,CCD)、雷射掃描螢光顯微鏡和雷射共聚焦掃瞄器等。其中的雷射共聚焦掃瞄器已發展為基因晶片的配套檢測系統。經過晶片掃描提取雜交信號之後,在數據分析之前,首先要扣除背景信號,進行數據檢查、標化和校正,消除不同實驗系統的誤差。對於簡單的檢測或科學實驗,因所需分析基因數量少,故直接觀察即可得出結論。若涉及大量基因尤其是進行表達譜分析時,就需要藉助專門的分析軟體,運用統計學和生物信息學知識進行深入、系統的分析,如主成分分析、分層聚類分析、判別分析和調控網路分析等。晶片數據分析結束並不表示晶片實驗的完成,由於基因晶片獲取的信息量大,要對呈數量級增長的實驗數據進行有效管理,需要建立起通行的數據儲存和交流平台,將各實驗室獲得的實驗結果集中起來形成共享的基因晶片資料庫,以便於數據的交流及結果的評估。
技術套用
在瘧原蟲研究中的套用
 基因晶片技術
基因晶片技術基因晶片技術也可用於比較不同種或同種不同株瘧原蟲的基因表達差異,Ganesan等利用晶片研究了惡性瘧原蟲Dd2和HB3株基因表達差異。Hall等比較了伯氏、夏氏、約氏和惡性瘧原蟲的基因表達差異。Gissot等利用晶片技術分析了紅內期不同時點惡性瘧原蟲3D7株及F12株之間表達譜的差異,並發現了影響配子體生成的候選基因。近年,基因晶片在瘧原蟲研究中的套用範圍進一步拓寬,研究內容涵蓋瘧原蟲新基因發現、疫苗靶標篩選、轉錄因子調控網路、瘧原蟲適應人體宿主機制、瘧原蟲比較基因組雜交分析、惡性瘧原蟲抗原變異分子機制以及瘧原蟲攻擊紅細胞機制等。總之基因晶片技術正逐步成為瘧原蟲分子生物學研究的有力工具。
在傳瘧按蚊研究中的套用
基因晶片在按蚊研究中最先用於探討按蚊對原蟲的抗性機制,在2003年,Kumar等利用基因晶片比較了對瘧原蟲易感和抗性的甘比亞按蚊基因表達的差異,發現甘比亞按蚊對瘧原蟲的抗性與活性氧(reactiveoxygenspecies,ROS)的產生和代謝關係密切。
按蚊對殺蟲劑的抗性問題日益突出,給瘧疾的預防和控制帶來困難,晶片技術的出現為人們認識按蚊殺蟲劑抗性的產生機制提供了新手段。2005年David和Vontas等設計出甘比亞按蚊基因晶片,比較了殺蟲劑敏感按蚊與抗性按蚊中230個殺蟲劑代謝相關基因的表達情況,結果發現谷胱甘肽S轉移酶(glutathione-S-transferase,GST)、細胞色素314A1(CYP314A1)、細胞色素P450s等基因與滴滴涕(dichloro-diphenyl-trichloroethane,DDT)抗性有關,擬除蟲菊酯抗性按蚊的P450、CYP325A3基因表達上調。2005年Dana和Marinotti等分別藉助cDNA和表達序列標籤(expressedsequencetag,EST)晶片,觀察血餐前後不同時間點甘比亞按蚊基因表達的變化,探討了按蚊對血細胞的消化、免疫反應以及卵的形成和運動的分子機制。2006年Dissanayake和Strode等利用基因晶片技術比較了甘比亞按蚊不同發育階段和不同組織的基因表達譜差異,發現約1/4基因的表達受發育調節。
此外,另有研究者分別利用基因晶片研究了按蚊與原蟲之間的相互作用機制、按蚊基因調控網路、按蚊唾液腺轉錄本、按蚊對原蟲的免疫應答等。總之,基因晶片技術的套用已顯著拓寬和提高了傳瘧按蚊的研究領域和研究水平。
在瘧疾動物模型研究中的套用
動物模型在人類瘧疾的病理學研究、疫苗開發和治療試驗等方面發揮著不可替代的作用,基因晶片技術與動物模型的結合,更進一步加深了人類在分子水平上對瘧疾的認識。2004年Sexton等利用基因晶片分析了鼠瘧原蟲轉錄組的變化,結果發現鼠感染原蟲後腦部400餘基因及脾臟600餘基因的轉錄發生了變化,這些變化反映了鼠紅細胞生成抑制、糖酵解和干擾素介導的免疫應答增強,從而找到瘧疾導致中樞神經系統症狀、貧血和乳酸增多症的可能機制。2005年Sarfo等利用cDNA晶片分析了趨化因子及其受體在約氏瘧原蟲感染小鼠腦部的表達情況,結果發現趨化因子RANTES(regulatedonactivation,normalTcellexpressedandsecreted)及其相應受體CCR1、CCR3和CCR5的mRNA在感染小鼠的腦部表達上調,參與了小鼠小腦的炎症介導、細胞降解和超微結構改變。2006年Delahaye等利用cDNA晶片比較了腦型瘧易感型與抵抗型小鼠腦部的基因表達差異,分析確定了可能與小鼠腦型瘧易感性有關的31個候選基因。
為探討病原體間的免疫協同作用,2005年Page等利用晶片分析了結核分支桿菌與約氏瘧原蟲混合感染及單獨感染時小鼠脾臟細胞的Ⅰ型免疫應答情況,發現混合感染組小鼠脾臟細胞γ干擾素(interferongamma,IFN2γ)和腫瘤壞死因子α(tumornecrosisfactoralpha,TNF2α)的生成水平顯著高於單獨感染組。結論認為結核分支桿菌可以增強Ⅰ型免疫應答來保護小鼠免受致死性鼠瘧損傷。2006年有研究者利用cDNA晶片分析了惡性瘧原蟲紅細胞膜蛋白Ⅰα(CDCRⅠα)的活化對人類基因表達的影響,結果提示CDCRⅠα可以激活多條免疫途徑,促進B細胞多克隆活化和凋亡延遲。從而找出了瘧疾流行區Burkitt's淋巴瘤危險性增加的潛在原因。