內容簡介
 siRNA
siRNARNA干擾現象是1990年由約根森(Jorgensen)研究小組在研究查爾酮合成酶對花青素合成速度的影響時所發現,為得到顏色更深的矮牽牛花而過量表達查爾酮合成酶,結果意外得到了白色和白紫雜色的矮牽牛花,並且過量表達查爾酮合成酶的矮牽牛花中查爾酮合成酶的濃度比正常矮牽牛花中的濃度低50倍。約根森推測外源轉入的編碼查爾酮合成酶的基因同時抑制了花中內源查爾酮合成酶基因的表達。
1992年,羅馬諾(Romano)和Macino也在粗糙鏈孢霉中發現了外源導入基因可以抑制具有同源序列的內源基因的表達。1995年,Guo和Kemphues線上蟲中也發現了RNA干擾現象。
1998年,安德魯·法厄(Andrew Z. Fire)等在秀麗隱桿線蟲(C.elegans)中進行反義RNA抑制實驗時發現,作為對照加入的雙鏈RNA相比正義或反義RNA顯示出了更強的抑制效果。從與靶mRNA的分子量比考慮,加入的雙鏈RNA的抑制效果要強於理論上1:1配對時的抑制效果,因此推測在雙鏈RNA引導的抑制過程中存在某種擴增效應並且有某種酶活性參與其中。並且將這種現象命名為RNA干擾。
2006年,安德魯·法厄與克雷格·梅洛(Craig C. Mello)由於在RNAi機制研究中的貢獻獲得諾貝爾生理及醫學獎。
原理作用
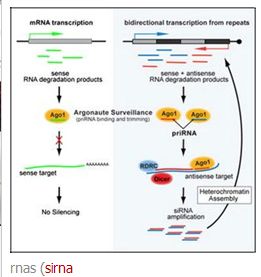
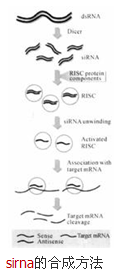
 siRNA
siRNARNA干涉(RNAi)在實驗室中是一種強大的實驗工具,利用具有同源性的雙鏈RNA(dsRNA)誘導序列特異的目標基因的沉寂,迅速阻斷基因活性。siRNA在RNA沉寂通道中起中心作用,是對特定信使RNA(mRNA)進行降解的指導要素。siRNA是RNAi途徑中的中間產物,是RNAi發揮效應所必需的因子。siRNA的形成主要由Dicer和Rde-1調控完成。由於RNA 病毒入侵、轉座子轉錄、基因組中反向重複序列轉錄等原因,細胞中出現了dsRNA,Rde-1(RNAi缺陷基因-1)編碼的蛋白質識別外源dsRNA,當dsRNA達到一定量的時候,Rde-1引導dsRNA與Rde-1編碼的Dicer(Dicer是一種RNaseIII 活性核酸內切酶,具有四個結構域:Argonaute家族的PAZ結構域,III型RNA酶活性區域,dsRNA結合區域以及DEAH/DEXHRNA解旋酶活性區)結合,形成酶-dsRNA複合體。Dicer 切割後形成siRNA,然後,在ATP的參與下,細胞中存在的一種RNA誘導的沉默複合體RNA-induced silencing complex RNAi干涉的關鍵步驟是組裝RISC和合成介導特異性反應的siRNA蛋白。siRNA併入RISC中,然後與靶標基因編碼區或UTR區完全配對,降解靶標基因,因此說siRNA只降解與其序列互補配對的mRNA。其調控的機制是通過互補配對而沉默相應靶位基因的表達,所以是一種典型的負調控機制。siRNA識別靶序列是有高度特異性的,因為降解首先在相對於siRNA來說的中央位置發生,所以這些中央的鹼基位點就顯得極為重要,一旦發生錯配就會嚴重抑制RNAi的效應。
siRNA的設計方法
關於設計siRNA以及siRNA設計對siRNA功能的影響,還沒有可靠的規律。雖然可以通過對mRNA實驗分析準確的找到一段基因的全部siRNA最佳作用位置,但這個過程十分耗時且昂貴,不推薦常規實驗使用。一種做法是每個目標序列設計3-4對siRNAs,實驗選擇較有效的siRNA。
siRNA設計大都根據Tuschl的實驗,要點如下:
1.目標基因開放閱讀框起始密碼下游75至100鹼基位置開始,尋找“AA”二連序列後的19個鹼基序列。(用非“AA”的“XY”序列後的19個鹼基序列同樣可以得到很好效果的siRNA)。 設計siRNA時不要針對5’和3’端的非編碼區。
2.分析獲得的序列,選擇GC比在40-55%之間的靶基因序列作為優選。
3.將潛在的序列和相應的基因組資料庫(人,或者小鼠,大鼠等等)進行比較,排除和其他編碼序列/EST同源的序列。例如使用BLAST等。
4.如果選擇shRNA,有報導顯示9個寡核苷酸的環是最有效的。
下面是另一個設計的版本,大致相同:
1. 從轉錄本(mRNA)的AUG起始密碼開始,尋找“AA”二連序列,並記下其3’端的19個鹼基序列,作為潛在的siRNA靶位點。有研究結果顯示GC含量在30%—50%左右的siRNA要比那些GC含量偏高的更為效。Tuschl等建議在設計siRNA時不要針對5’和3’端的非編碼區(untranslated regions,UTRs),原因是這些地方有豐富的調控蛋白結合區域,而這些UTR結合蛋白或者翻譯起始複合物可能會影響siRNP核酸內切酶複合物結合mRNA從而影響siRNA的效果。
2. 將潛在的序列和相應的基因組資料庫(人,或者小鼠,大鼠等等)進行比較,排除那些和其他編碼序列/EST同源的序列。例如使用BLAST( www.ncbi.nlm.nih.gov/BLAST/)
3. 選出合適的目標序列進行合成。通常一個基因需要設計多個靶序列的siRNA,以找到最有效的siRNA序列。
一個完整的siRNA實驗應該有負對照,作為負對照的siRNA應該和選中的siRNA序列有相同的組成,但是和mRNA沒有明顯的同源性。通常的做法是將選中的siRNA序列打亂,同樣要檢查結果以保證它和其他基因沒有同源性。 如果計畫合成siRNA,那么可以直接提供以AA打頭的21個鹼基序列,廠家會合成一對互補的序列。注意的是通常合成的siRNA是以3’dTdT結尾,如果要UU結尾的話通常要特別說明。有結果顯示,UU結尾和dTdT結尾的siRNA在效果上沒有區別。因為這個突出端無需和靶序列互補。
現在Qiagen(http://www.qiagen.com/)公司的網站上有完整的siRNA 的設計方法,可以自動設計並生成多個備選序列,並且可以自動連線到基因組資料庫進行檢索比對。
siRNA的製備
體外製備
1.化學合成
許多國外公司都可以根據用戶要求提供高質量的化學合成siRNA。主要的缺點包括價格高,定製周期長,特別是有特殊需求的。由於價格比其他方法高,為一個基因合成3—4對siRNAs 的成本就更高了,比較常見的做法是用其他方法篩選出最有效的序列再進行化學合成。
最適用於:已經找到最有效的siRNA的情況下,需要大量siRNA進行研究
不適用於:篩選siRNA等長時間的研究,主要原因是價格因素
2.體外轉錄
以DNA Oligo為模版,通過體外轉錄合成siRNAs,成本相對化學合成法而言比較低,而且能夠比化學合成法更快的得到siRNAs。不足之處是實驗的規模受到限制,雖然一次體外轉錄合成能提供足夠做數百次轉染的siRNAs,但是反應規模和量始終有一定的限制。而且和化學合成相比,還是需要占用研究人員相當的時間。值得一提的是體外轉錄得到的siRNAs毒性小,穩定性好,效率高,只需要化學合成的siRNA量的1/10就可以達到化學合成siRNA所能達到的效果,從而使轉染效率更高。
最適用於:篩選siRNAs,特別是需要製備多種siRNAs,化學合成的價格成為障礙時。
不適用於:實驗需要大量的,一個特定的siRNA。長期研究。
3.用RNase III 消化長片斷雙鏈RNA製備siRNA
其他製備siRNA的方法的缺陷是需要設計和檢驗多個siRNA序列以便找到一個有效的siRNA。而用這種方法——製備一份混合有各種siRNAs “混合雞尾酒” 就可以避免這個缺陷。選擇通常是200—1000鹼基的靶mRNA模版,用體外轉錄的方法製備長片斷雙鏈dsRNA ,然後用RNase III (or Dicer) 在體外消化,得到一種siRNAs“混合雞尾酒”。在除掉沒有被消化的dsRNA後,這個siRNA混合物就可以直接轉染細胞,方法和單一的siRNA轉染一樣。由於siRNA混合物中有許多不同的siRNAs,通常能夠保證目的基因被有效地抑制。
dsRNA消化法的主要優點在於可以跳過檢測和篩選有效siRNA序列的步驟,為研究人員節省時間和金錢(注意:通常用RNAse III通常比用Dicer要便宜)。不過這種方法的缺點也很明顯,就是有可能引發非特異的基因沉默,特別是同源或者是密切相關的基因。現在多數的研究顯示這種情況通常不會造成影響。
最適用於:快速而經濟地研究某個基因功能缺失的表型
不適用於:長時間的研究項目,或者是需要一個特定的siRNA進行研究,特別是基因治療
體內表達
前面的3種方法主要都是體外製備siRNAs,並且需要專門的RNA轉染試劑將siRNAs轉到細胞內。而採用siRNA表達載體和基於PCR的表達框架則屬於:從轉染到細胞的DNA模版中在體內轉錄得到siRNAs。這兩種方法的優點在於不需要直接操作RNA。
4. siRNA表達載體
多數的siRNA表達載體依賴三種RNA聚合酶III 啟動子(pol III)中的一種,操縱一段小的髮夾RNA(short hairpin RNA, shRNA)在哺乳動物細胞中的表達。這三類啟動子包括大家熟悉的人源和鼠源的U6啟動子和人H1啟動子。之所以採用RNA pol III啟動子是由於它可以在哺乳動物細胞中表達更多的小分子RNA,而且它是通過添加一串(3到6個)U來終止轉錄的。要使用這類載體,需要訂購2段編碼短髮夾RNA序列的DNA單鏈,退火,克隆到相應載體的pol III 啟動子下游。由於涉及到克隆,這個過程需要幾周甚至數月的時間,同時也需要經過測序以保證克隆的序列是正確的。
siRNA表達載體的優點在於可以進行較長期研究——帶有抗生素標記的載體可以在細胞中持續抑制靶基因的表達,持續數星期甚至更久。
病毒載體也可用於siRNA表達,其優勢在於可以直接高效率感染細胞進行基因沉默的研究,避免由於質粒轉染效率低而帶來的種種不便,而且轉染效果更加穩定。
最適用於:已知一個有效的siRNA序列,需要維持較長時間的基因沉默。
不適用於:篩選siRNA序列(其實主要是指需要多個克隆和測序等較為費時、繁瑣的工作)。
5. siRNA表達框架
siRNA表達框架(siRNA expression cassettes,SECS)是一種由PCR得到的siRNA表達模版,包括一個RNA pol III啟動子,一段髮夾結構siRNA,一個RNA pol III終止位點,能夠直接導入細胞進行表達而無需事前克隆到載體中。和siRNA表達載體不同的是,SECs不需要載體克隆、測序等頗為費時的步驟,可以直接由PCR得到,不用一天的時間。因此,SECs成為篩選siRNA的最有效工具,甚至可以用來篩選在特定的研究體系中啟動子和siRNA的最適搭配。如果在PCR兩端添加酶切位點,那么通過SECs篩選出的最有效的siRNA後,可以直接克隆到載體中構建siRNA表達載體。構建好的載體可以用於穩定表達siRNA和長效抑制的研究。
這個方法的主要缺點是①PCR產物很難轉染到細胞中(晶賽公司的Protocol可以解決這一問題)②不能進行序列測定,PCR和DNA合成時可能差生的誤讀不能被發現導致結果不理想。
最適用於:篩選siRNA序列,在克隆到載體前篩選最佳啟動子
不適用於:長期抑制研究。(如果克隆到載體後就可以了)
主要套用
RNAi在基因沉默(silent gene)方面具有高效性和簡單性,所以是基因功能研究的重要工具。
大多數藥物屬於標靶基因(或疾病基因)的抑制劑,因此RNAi 模擬了藥物的作用,這功能丟失(LOF)的研究方法比傳統的功能獲得(GOF)方法更具優勢。因此, RNAi 在今天的製藥產業中是藥物靶標確認的一個重要工具。同時,那些在靶標實驗中證明有效的siRNA/shRNA本身還可以被進一步開發成為RNAi藥物。
在藥物標靶發現和確認方面,RNAi技術已獲得了廣泛的套用。生物技術公司或製藥公司通常利用建立好的RNAi文庫來引入細胞,然後通過觀察細胞的表型變化來發現具有功能的基因。如可通過RNAi文庫介導的腫瘤細胞生長來發現能抑制腫瘤的基因。一旦所發現的基因屬於可用藥的靶標(如表達的蛋白在細胞膜上或被分泌出細胞外),就可以針對此靶標進行大規模的藥物篩選。此外,被發現的靶標還可用RNAi技術在細胞水平或動物體內進一步確認。
在疾病治療方面,雙鏈小分子RNA或siRNA已被用於臨床測試用於幾種疾病治療,如老年視黃斑退化、肌肉萎縮性側索硬化症、類風濕性關節炎、肥胖症等。在抗病毒治療方面,帕金森病等神經系統疾病已經開始初步採用RNA干擾療法。腫瘤治療方面也已經取得了一些成果。
研究策略
siRNA 研究策略與方法.pdf