簡介
國際人類基因組單體型圖計畫(The International HapMap Project)是繼國際人類基因組計畫(The Human Genome Project,HGP)之後人類基因組研究領域的又一個重大研究計畫。HapMap計畫是一個多國參與的合作項目,旨在確定和編目人類遺傳的相似性和差異性。利用HapMap獲得的信息,研究人員將能夠發現與人類健康、疾病以及對藥物和環境因子的個體反應差異相關的基因。項目由來自日本、英國、加拿大、中國、奈及利亞和美國的科學家和資助機構合作完成,所產生的全部數據將免費向公眾開放。
發展
HapMap計畫起始於2002年,由美、加、中、日、英、奈及利亞等國研究機構發起、參與及完成。中國科學家們由中科院北京基因組研究所牽頭,承擔3號、21號和8號染色體短臂單體型圖的構建,約占總計畫的10%。項目共取樣270個正常個體,其中歐洲30個三聯家系,亞洲45箇中國人,45個日本人,非洲30個三聯家系。2005年10月26日,由國際協作組總負責人,現任美國NIH負責人Francis Collins向全世界宣布了一個擁有數億數據的人類基因組單體型圖的成功構建,以及一個更精細的遺傳圖譜即將完成。至此hapmap一期計畫證實宣告完成。一期計畫共成功分型100多萬個多態性位點,也稱單核苷酸多態性(SNP)位點,全基因組平均3kb一個SNP位點。由於染色體塊狀結構,也即連鎖不平衡的存在,一期數據可以捕獲大部分基因組上的遺傳差異信息。HapMap計畫的第一階段任務完成後,國際協作組委託Perlegen Sciences 完成第二階段擴大SNP分型密度的任務。2007年10月18日,國際協作組在Nature上發表了根據第二階段數據構建的人類基因組的第二代HapMap。至HapMap 二期共發現了超過一千萬的人類基因組的SNPs,完成了約310萬SNPs(≥5%)在270個樣品中的分型反應。這些SNPs約占預測的遺傳變異的25%~35%,並使第二代HapMap的解析度達到平均不到1kb一個SNP,比預定計畫超過100%,準確度達到99.8%。二期數據密度約為一期數據的3倍,構建起一張精度更高信息更完整的多人種遺傳多態圖譜。可以說正是這些遺傳多態位點的存在,在遺傳體質上,你我之間有了個體差異,不同人種之間有了種群差異。
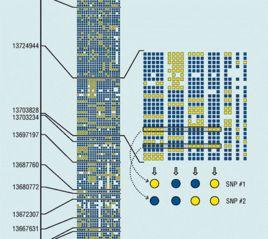
HapMap計畫建立了人類全基因組遺傳多態圖譜,依據這張圖譜我們可以進一步研究基因組的結構特點以及SNP位點在人群間的分布情況,為群體遺傳學的研究提供數據,為遺傳性疾病致病基因在基因組上的定位提供高密度的SNP位點。HapMap的構建分為三個步驟:(a)在多個個體的DNA樣品中鑑定單核苷酸多態性(SNPs);(b)將群體中頻率大於1%的那些共同遺傳的相鄰SNPs組合成單體型;(c)在單體型中找出用於識別這些單體型的標籤SNPs。通過對圖中的三個標籤SNPs進行基因分型,可以確定每個個體擁有哪一個單體型。
二期數據的在密度上大大提高,給各種基因組精細研究提供了更大的支持。
結合群體遺傳學的研究手段,我們可以更加深入地去觀察和研究使我們之所以成為我們的基因組。重組率推算提供了我們一張基因組進化痕跡圖;連鎖不平衡的計算給了我們一張基因組塊狀連鎖結構圖;種群差異FST的計算讓我們看到一張種群間基因組結構差異圖;SNP的雜合情況告訴我們人類基因組上受到選擇的區域或區域內的基因;利用SNP位點向兩邊延伸的長度差異情況,我們可以觀察到一些基因組上近期正在進行的選擇事件,甚至是當前正在悄悄進行中的進化,因為新產生的突變位點傳代較少,它和周圍位點的連鎖情況受重組事件的影響較小,另一方面優勢突變也會因選擇壓力的存在使周圍的重組受到影響…...當然這些不同的指標中也隱藏了人類成長過程中的一些信息,例如遷徙,戰爭,災難,繁盛等各種對基因組遺傳多態性產生影響的歷史事件。
高密度的SNP位點,為全基因組相關性分析(GWA)提供了可能,以往遺傳學上定位基因的使用較多的工具是微衛星,這一套新產生的SNP位點彌補了微衛星在基因組上分布不夠均勻,以及密度不夠高的缺點,SNP聯合微衛星將使目的基因的精細定位更加可行。目前已經有很多致病基因藉助SNP數據得到定位。另外,根據SNP在基因的不同功能元件中的分布情況和基因在細胞中的表達情況,我們可以研究基因上的不同元件序列是如何控制蛋白表達進而影響個體表型的。
伴隨著HapMap二期數據的產出和各種實驗技術的進一步發展以及更加大量的基因組序列數據加入到人類的知識庫中,我們將能更加完整更加深入和更加正確地認識我們自己,認識自然界的奧秘,揭示生命的本質。