啟動子區的基本結構
啟動子是一段位於結構基因5'端上游區的DNA序列,能活化RNA聚合酶,使之與模板DNA準確地相結合併具有轉錄起始的特異性。因為基因的特異性轉錄取決於酶與啟動子能否有效地形成二元複合物,故RNA聚合酶如何有效地找到啟動子並與之相結合是轉錄起始過程中首先要解決的問題。有實驗表明,對許多啟動子來說,RNA聚合酶與之相結合的速率至少比布朗運動中的隨機碰撞高100倍。
轉錄的起始是基因表達的關鍵階段,而這一階段的重要問題是RNA聚合酶與啟動子的相互作用:啟動子的結構影響了它與RNA聚合酶的親和力,從而影響了基因表達的水平。
為什麼RNA聚合酶能夠僅在啟動子處結合呢?顯然啟動子處的核苷酸順序具有特異的形狀以便與RNA聚合酶結合,就好像酶與其底物的結構相恰恰適合一樣。將100個以上啟動子的順序進行了比較,發現在RNA合成開始位點的上游大約10bp和35bp處有兩個共同的順序,稱為-10和-35序列。這兩個序列的共同順序如下,-35區“AATGTGTGGAAT”,-10區“TTGACATATATT”。大多數啟動子均有共同順序(consensus sequence),只有少數幾個核苷酸的差別。
轉錄單元
轉錄單元(transcription unit) 是一段從啟動子開始至終止子(terminator)結束的DNA序列,RNA聚合酶從轉錄起點開始沿著模板前進,直到終止子為止,轉錄出一條RNA鏈。在細胞中,一個轉錄單元可以是一個基因,也可以是幾個基因。
轉錄起點
轉錄起點是指與新生RNA鏈第一個核苷酸相對應DNA鏈上的鹼基,研究證實通常為一個嘌呤。常把起點前面,即5’末端的序列稱為上游(upstream),而把其後而即3’未端的序列稱為下游(downstream)。在描述鹼基的位置時,一般用數字表示,起點為+1,下遊方向依次為+2、+3……,上遊方向依次為-1、-2、-3……。
啟動子區
啟動子區是RNA聚合酶的結合區,其結構直接關係到轉錄的效率。關於其結構特點,Pribnow設計了一個實驗,他把RNA聚合酶全酶與模板DNA結合後,用DNase l水解DNA,然後用酚抽提,沉澱純化DNA後得到一個被RNA聚合酶保護的DNA片段,約有41~44個核苷酸對。他先後分離了fd噬菌體、T7噬菌體的A2及A3啟動子、h噬σ菌體的PR啟動子及大腸桿菌乳糖操縱子的UV5啟動子等5段被酶保護的區域,並進行了序列分析,以後又有人做了50多個啟動子的序列分析後發現,在被保護區內有一個由5個核苷酸組成的共同序列,是RNA聚合酶的緊密結合點,稱為Pribnow區(Pribnow box),這個區的中央大約位於起點上游10bp處,所以又稱為-10區。
許多原核生物都含有這兩個重要的啟動子區:RNA聚合酶同啟動子結合的區域稱為啟動子區。將各種原核基因同RNA聚合酶全酶結合後,用DNase I水解DNA,最後得到與RNA聚合酶結合而未被水解的DNA片段,這些片段有一個由5個核苷酸(TATAA)組成的共同序列,以其發現者的名字命名為Pribnow框(Pribnowbox),這個框的中央位於起點上游10bp處,所以又稱-10序列(-10 sequence),後來在-35 bp處又找到另一個共同序列(TTGACA)。Hogness等在真核基因中又發現了類似Pribnow框的共同序列,即位於-25~-30 bp處的TATAAAAG,也稱TATA框(TATAbox)。TATA框上游的保守序列稱為上游啟動子元件(upstream promoter element,UPE)或上游激活序列(uptreamactivatingsequence,UAS)。另外在-70~-78 bp處還有一段共同序列CCAAT,稱為CAAT框(CAAT box)。
在真核生物基因中,Hogness等先在珠蛋白基因中發現了類似Pribrow區的Hogness區(Hogness box),這是位於轉錄起始點上游-25~-30 bp處的共同序列似TAAA,也稱為TATA區。另外,在起始位點上游-70~-78 bp處還育另一段共同序列CCAAT,這是與原核生物中-35bp區相對應的序列.稱為CAAT區(CAAT box)。
-10位區和-35位區
提純被保護的片段後卻發現,RNA聚合酶並不能重新結合或並不能選擇正確的起始點,表明在保護區外可能還存在與RNA聚合酶對啟動子的識別有關的序列。果然,科學家不久就從噬菌體的左、右啟動子PL及PR和SY40啟動子的-35 bp附近找到了另一段共同序列:TTGACA。經過數年的努力,分析了46個大腸桿菌啟動子的序列以後.確證絕大部分啟動子都存在這兩段共同序列,即位於-10bp處(……T89A89T50A65A100……)的TATA區和-35 bp處(……T85T83G81A61C69A52……)的TTGACA區。現已查明,-10位的TATA區和-35位的TTGACA區是RNA聚合酶與啟動子的結合位點,能與σ因子相互識別而具有很高的親和力。
原核生物中-10區同-35區之間核苷酸數目的變動會影響基因轉錄活性的高低,強啟動子一般為17±1 bp,當間距小於15 bp或大於20 bp時都會降低啟動子的活性。
以下是核苷出現的機率:
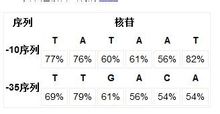 核苷出現的機率表格
核苷出現的機率表格在真核基因中,有少數基因沒有TATA框。沒有TATA框的真核基因啟動子序列中,有的富集GC,即有GC框;有的則沒有GC框。GC框位於-80~-110bp處的GCCACACCC或GGGCGGG序列。
TATA框的主要作用是使轉錄精確地起始;CAAT框和GC框則主要是控制轉錄起始的頻率,特別是CAAT框對轉錄起始頻率的作用更大。如在TATA框同相鄰的UPE之間插入核苷酸,也會影響轉錄使之減弱。
-10序列又稱為Pribnow盒(原核生物)。在真核生物中相應的序列位於-35bp處,稱為TATA盒,又稱為Goldberg-Hognessbox,是RNA聚合酶Ⅱ的結合部位。-10和-35這兩個部位都很重要:【1】RNA聚合酶能和-35和-10序列中的鹼基和DNA主鏈中的磷酸基相接觸;【2】離開共同順序較遠的啟動子的活性亦較弱;【3】最重要的是,破壞啟動子功能的突變中有75%都是改變了共同順序中的鹼基,其餘25%亦為離共同順序較近的。-35和-10序列相距約20bp,即大致是雙螺鏇繞兩圈的長度。因為這兩個結合區是在DNA分子的同一側面,可見此酶是結合在雙螺鏇的一面。可以想像,它能"感覺到每個結合區的溝底中鹼基所產生的特異形狀。"
原核生物亦有少數啟動子缺乏這兩個序列(-35和-10)之一。在這種情況下,RNA聚合酶往往不能單獨識別這種啟動子,而需要有輔助蛋白質的幫助。可能是這些蛋白質因子與鄰近序列的反應可以彌補啟動子的這個缺陷。
在真核生物中,在轉錄起始位點上游70-80bp處有CAAT順序,也稱為CAAT盒。這一順序也是比較保守的共同順序:GCCTCAATCT。RNA聚合酶Ⅱ可以識別一順序。在對家兔β珠蛋白基因CAAT順序的研究中發現,用人工方法誘導CAAT順序發生突變使家兔β珠蛋白基因的轉錄水平降低。
啟動子中的-10和-35序列是RNA聚合酶所結合和作用必需的順序。但是附近其他DNA順序也能影響啟動子的功能。例如,在核糖體RNA合成的起始位點的上游50到150核苷酸之間的順序就是對啟動子的完全活性所必需的。如果這一段DNA順序缺失並由其他外來DNA所取代(例如克隆在質粒DNA中的rRNA基因),則轉錄起始的頻率將降低10倍。同樣,在其他情況下,遠隔部位的富有AT的DNA順序被認為能增進轉錄起始的頻率。有時候上游順序可以是某些能直接激活RNA聚合酶的"激活蛋白"的結合部位。但是,上游順序往往有另外的功能。例如上游順序可以吸引拓撲異構酶,後者可導致結合的局部產生有利於轉錄起始的超螺鏇狀態。上游順序所引起的DNA結構的微細變化可能在雙螺鏇上被傳導到相當遠的距離,因此上游順序的變化可以影響到-10和-35區的DNA結構細節。
啟動子功能變異的疾病
以下是從人類孟德爾遺傳學(OMIM)證實與啟動子故障有關,不論是因啟動子序列直接突變或是轉錄因子或轉錄共激發因子的突變。而多種癌症都沒有列下是因為從染色體易位產生嵌合基因:
哮喘 β地中海貧血 魯賓斯坦泰比綜合症
要留意的是在病原學上大部份的疾病都是異質的,而在分子層面上一種疾病往往是指多種疾病,縱然它們的病徵及治療方法一致。疾病對治療有不同的反應,是因背後分子源頭的差異,這會是藥物遺傳學的範疇。