性能及歷史
蛋白質資料庫(HPDB),建於2005年5月, 動態展示生物大分子立體結構,滑鼠點擊放大分子結構、原子定位、測定原子之間距離,可用於教學或科研。 服務對象是能夠熟練使用中文的生命科學、醫學、藥學、農學、林學等領域的大中專學生、教師及科技工作者 。分子結構特徵描述採用漢語,同時提供英文原文以供考證。 對於善於使用英文的讀者,我們提倡直接訪問RCSB PDB,一來可以減少網路擁擠,二來可以減少由於 HPDB 的翻譯不妥帶來的不便。
蛋白質資料庫(HPDB)對每個蛋白質分子結構說明部分做了中文翻譯(最新加入資料庫的分子除外),內容包括分子結構定性描述、樣品的來源、表達載體、宿主、化學分析方法、分子結構組成成分等。 這些信息並同蛋白質分子結構數據存儲於資料庫, 因此 HPDB 支持中文查詢。
蛋白質資料庫(HPDB)雖然翻譯了“分子結構說明”部分,但為了保證數據的可靠性和準確性,HPDB對一級結構序列及大分子結構坐標數據等未做任何改動,資料庫保持 RCSB PDB 核實後的原始實驗數據檔案,並保持 PDB 檔案格式和蛋白質分子編號 。
布魯克海文蛋白質資料庫(TheBrookHavenProteinDataBank,PDB)是由美國布魯克海文國家實驗室所維護的關於生物大分子三維結構的數據檔案,其內容包括生物大分子的原子坐標、參考文獻、1級和2級結構信息,也包括了晶體結構因數以及NMR實驗數據。PDB由美國國家科學基金等組織提供資助,對全球的科研工作者、教育工作者以及學生等提供免費服務。
PDB創立於1973年,到了90年代,PDB中的數據開始逐步發展豐富起來。據統計,從1992—1996年該庫收集的生物大分子結構的數目分別是1007、1727、2921、3821和4707,平均每年遞增50%。到1998年4月8日為止,該庫共收集了7429個原子坐標的入口檔案,1739個結構因數檔案,429個NMR抑制檔案。PDB中主要收集蛋白質的結構信息,也包括了少量的核酸及糖的三維結構。獲得信息的實驗技術主要為X線衍射技術以及NMR實驗技術 。
檔案結構
在蛋白質晶體結構資料庫PDB中,各大分子結構是以分立的檔案形式記錄的,這些檔案被稱作PDB的入口檔案(entry)。1個檔案只反映某個大分子結構的信息。每個大分子結構通過唯一的ID碼(4位代碼)來識別。早期的入口檔案檔案名稱後綴為“.pdb”,1種大分子對應1個檔案,如:阿比西尼亞捲心菜(ABYSSINIAN CABBAGE SEED)種子蛋白的ID碼是1CRN,其入口檔案名稱為1CRN.pdb。1997年以後,每1種生物大分子有1組(3個)相關檔案與之對應,它們是:全文檔案、書目檔案和圖形檔案,例如,抗生素MINORCOATPROTEIN的ID碼是1G3P,它的3個相關檔案分別為1G3P.full(全文檔案)、1G3P.biblio(書目檔案)、1G3P.gif(圖形檔案);免疫球蛋白(IMMUN-O GLOBULIN)的ID碼為1AP2,它的3個相關檔案分別為1AP2.full(相當於原來的.pdb檔案),1AP2.biblio,1AP2.gif,等等。
每1個PDB入口檔案包含有標題部分、注釋部分、1級結構、異質、2級結構、連通性注釋、各種特性、結晶學、坐標變換、原子坐標、化學連線、薄記等12個部分。檔案中每1行被稱作1條記錄,也被稱作入口(entry),可理解為記錄入口。每行包括80列,每個記錄入口的最後1個字元是1個行結束符。PDB檔案也可以被看作記錄類型(recordtype)的集合。它和一般的關係資料庫概念不同。在關係資料庫的庫檔案中,每條記錄由不同數據類型和數據格式的若干欄位組成,所有記錄的欄位結構都是相同的。而在PDB檔案中,包含眾多的記錄類型,每類記錄都有不同的格式。
基於記錄類型在1個PDB入口檔案中出現的次數可將1組記錄劃分成以下6類之一:
•single:單次記錄型。如HEADER、END、CRYST1……,在1個檔案中僅出現1次,沒有接續部分。
•singlecontinued:單次接續型。如AUTHOR、CAVEAT、COMPND……,在1個檔案中概念性地存在1次,其內容超過1行,可表示在後續行中,這些後續行包括1個接續指示欄位。
•multiple:多次記錄型。如ATOM、CONECT、HELIX……,在1個檔案中出現多次,在這類記錄類型中信息以列表的形式出現。
•multiplecontinued:多次接續型。例如FORMUL、HETATM、HETNAM,在1個入口檔案中概念性地存在多次,每條記錄內容超過1行的部分可表示在後續行中,這些後續行包括1個接續指示欄位。
•grouping:用來作為其他記錄類別的分組標誌記錄型。如:ENDMDL、MODEL、TER。
•other:其他記錄類型,如:JRNL定義坐標系列的文獻引用,REMARK表示一般注釋。每個記錄類型被固定列數分割成若干欄位,欄位應包含數據類型、欄位名和欄位定義。沒有被定義的列應留空。
大分子結構
一級結構
在生物化學上定義1級結構為蛋白質分子中胺基酸殘基的排列順序。1級結構中的胺基酸是蛋白質最基本的結構單位,標準的胺基酸有20多種,在1個胺基酸中含有氨基—NH2和羧基—COOH,若氨基中失去1個H原子,羧基中失去OH基團,就形成了殘基。2個胺基酸可以脫水縮和成肽,形成肽鍵和穩定的肽平面。相鄰2個胺基酸殘基以肽鍵相連線,依次連線下去即構成了1級結構肽鏈。
在PDB入口檔案中,蛋白質1級結構部分主要描述生物大分子每條鏈中胺基酸的排列順序。該部分包含有DBREF、SEQADV、SEQRES、MODRES等4種記錄,其中SEQRES記錄對胺基酸殘基有序排列進行了描述。例如,在1ROG(組織相容性抗原HLA-B*2705)入口檔案中共有16條SEQRES記錄,分為A、B2個鏈,每行為1條記錄,按排列順序列出胺基酸殘基,一行一行連貫下去,就組成了由GLY、SER、HIS,……胺基酸殘基順序連線而成的肽鏈。下例是由1ROG.pdb檔案中摘錄的部分內容:
如前所述,每行是1條記錄,每行中第1個欄位是記錄名“SEQRES”。第2個欄位是1個整數,表示記錄在當前鏈中的序號。第3個欄位是鏈的標識符,本例有A和B2個鏈,如果僅有1個鏈,則該欄位域為空。第4個欄位是1個整數,表示本鏈中胺基酸殘基數。第5~17欄位是胺基酸殘基序列,每個欄位是1個胺基酸名字。
異質
在PDB檔案的異質(heterogen)部分包含了對非標準胺基酸殘基的說明。這一部分包含HET、HETNAM、HETSYN、FORMUL等4種記錄。HET記錄描述了已給出坐標的非標準胺基酸,如:溶性分子、輔基、鐵等,同時也描述未知化學名稱的異質。在1G3P入口檔案里,有關HET的第1條記錄為:
HET TRO 21 15
這裡“HET”為記錄名稱,TRO為HET標識符,21為順序編號,15為HETATM記錄中本組群出現的次數,也就是說,本記錄描述了順序編號為21的非標準胺基酸TRO在坐標入口HETATM記錄中出現15次。HETNAM記錄描述了具有給定非標準胺基酸標識符的化合物的化學名稱。例如,1G3P檔案中HETNAM記錄之一為:
HETNAM SO4 SULFATE ION
描述了HET標識符為SO4的化合物的化學名稱為SULFATEION。FORMUL記錄描述了非標準組群的化學表示式及其所帶的電荷數。
二級結構
2級結構是指多肽鏈的主鏈骨架以肽平面為單位盤曲、摺疊而形成的構像。2級結構包括了3種情況:
•α-螺旋
•β-片層
•β-轉角。
在β-片層中,肽鍵平面摺疊成鋸齒狀,相鄰2個肽鍵平面之間的夾角呈110°角。在PDB入口檔案中,描述蛋白質2級結構的有HELIX、SHEET、TURN3種記錄。HELIX記錄用於描述分子中α-螺旋結構的位置。給出螺旋的名稱和編號,標示出螺旋開始和結束處的殘基,以及總長度。SHEET記錄用於描述分子中β-片層結構的位置。記錄格式與HELIX類似。TURN記錄描述摺疊和轉角。
從PDB的Web主頁(http://www.rcsb.org/pdb/index.html)可以按大分子的4位代碼查找。打開了某個蛋白質大分子的網頁後,可以查看總體信息、三維結構、序列細節等,還可以下載PDB入口檔案。例如,1ROG的胺基酸序列和2級結構如下:
這裡,每個胺基酸殘基用單個字母表示,2級結構(2、4、6、8行)用H表示螺旋,B表示隔離的β橋上的殘基,E表示擴展的β鏈,G表示310螺旋,I表示pi螺旋,T表示氫鍵轉角,S表示彎曲,等。
連線部分
這一部分描述了有關二硫鍵及其他一些化學連線情況,描述化學連線的記錄有SSBOND、CONECT、LINK、HYDBND、CISPEP等5種。SSBOND記錄描述了蛋白質和多肽結構中的二硫鍵。CONECT記錄表示了其他記錄未能表示出的原子間關聯狀態。例如,在1G3P檔案中,有關CONECT的第1條記錄為:CONECT4948299這裡“CONECT”為記錄名稱,其後內容表示在ATOM或HETATM記錄中的第48位原子和第299位原子分別與第49位原子有成鍵關係。LINK記錄詳細描述了在1級結構中不能明確的殘基間的關係,它實質上是上面介紹的CONECT記錄的1個補充。HYDBND記錄描述了原子間形成的氫鍵。
坐標章節
坐標章節主要記錄了原子的坐標,相關的記錄有:ATOM、HETATM、MODEL與ENDMDL。ATOM記錄按照從氨基到羧基的順序給出標準胺基酸殘基的各組成元素的空間坐標,從生物化學角度,我們可以對標準胺基酸殘基空間結構中的各原子間的連線情況進行描述。以在ATOM記錄中位於肽鏈第1位的標準胺基酸殘基ALA為例:ALA殘基中所包含的元素及其排列序號為1N2CA3C4O5CB。其中:
2CA中的A=alpha(α);
5CB中的B=beta(β)。
通過以上描述,確定了單個殘基的空間位置及各原子相互關係。根據肽鏈中相鄰2個殘基發生脫水縮合形成肽鍵及穩定的肽平面這一性質,我們可以確定1級結構中相鄰的2個胺基酸殘基間的關係,具體描述為前1個殘基結構中的碳原子(CA)與後1個殘基結構中的氮原子(N)間形成肽平面,以此類推,就會描繪出1級結構中各個相鄰殘基間的關係。
在1G3P檔案中,有關第1個殘基ALA的ATOM記錄為:
“ATOM”為記錄名稱,上例第1條記錄描述了殘基ALA中的氮元素(N)的x、y、z坐標值分別為-10.684、7.361和121.696,所占空間為1.00,溫度係數為17.19,元素符號為N,其他ATOM記錄針對殘基ALA包含的其他元素進行了描述。在1G3P檔案中用同樣方法描述出其餘217個處於不同空間位置的胺基酸殘基的原子坐標。HETATM記錄描述了組成非標準胺基酸殘基的元素(非標準胺基酸殘基名稱已在HET記錄中給出定義)的空間位置坐標。它的記錄方式與ATOM記錄一致。TER記錄標記出ATOM記錄的終止位。MASTER記錄是對以上各記錄的總結。下例中所列數字分別代表了記錄REMARK、“0”、HET、HELIX、SHEET、TURN、SITE、坐標變換、原子記錄、TER、CONECT、SEQRES的記錄總數。例:MASTER25802213006188912817END記錄表明了文章的結束,記錄格式為END。
可視化
生物大分子三維結構可視化
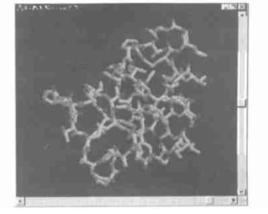
 蛋白質資料庫
蛋白質資料庫根據以上介紹可知,在PDB資料庫中,生物大分子的結構是通過特定的記錄格式,以原子空間坐標值和對於其連線形式、連線順序等的描述來表示的。通過特定的瀏覽器,如Rasmol,可基於PDB檔案實現對於大分子三維結構的可視化。Rasmol是1個分子現象程式。可嵌入Web瀏覽器中運行,用於通過超連結,打開網際網路上的pdb檔案,也可在Windows環境下,或在Mac和Unix環境下單機操作。圖是運行Rasmenu.exe後出現的主選單視窗,在主選單視窗後面,還同時出現1個分子圖形圖像顯示視窗。在主選單視窗中打開1個pdb檔案,其三維結構圖形就在第2個視窗中顯示出來。可在主選單中選擇顯示形式,如:線狀、棒狀、棒球狀、帶狀,等等,以及選擇顏色的區分方式等等。用滑鼠操作,可從不同角度去觀察分子三維結構,就像你身臨現場,從不同角度去觀察1件玲瓏剔透的牙雕藝術品一樣,十分精美壯觀 。