概述
 DNA條形碼
DNA條形碼DNA條形碼是指生物體內能夠代表該物種的,標準的、有足夠變異的、易擴增且相對較短的DNA片段。在發現一種未知物種或者物種的一部分時,研究人員便描繪其組織的DNA條形碼,而後與國際資料庫內的其他條形碼進行比對。如果與其中一個相匹配,研究人員便可確認這種物種的身份。
DNA條形碼已經成為生態學研究的重要工具,不僅用於物種鑑定,同時也幫助生物學家進一步了解生態系統內發生的相互作用。
條形碼位置
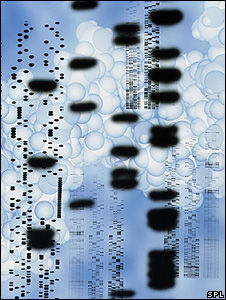 條形碼位置
條形碼位置DNA條形編碼,根據對一個統一的目的基因DNA序列的分析,把分析的結果儲存到計算機里,一旦需要,即可把要分析的物種的目的基因DNA序列與預存的數據進行比對,達到物種鑑定的目的。
這種新檢測法將注意力放線上粒體DNA上。與細胞核的DNA相比,線粒體DNA進化的更快。在兩個種群停止雜交繁殖後,一些特殊的變化將會線上粒體DNA中積累起來,兩個種群的該DNA將會出現差別,因此生物學工作者就能夠分辨出兩個很相近的生物是否是不同種。
為了讓條形碼資料庫成為一種經濟有效的工具,目前主要研究在所有生物當中都存在的一個基因———細胞色素氧化酶I(COI,cytochromecoxidaseI)基因的一部分。這部分DNA序列能夠區分出物種差異,就像物種的“身份證”編號。比對方法
 比對方法
比對方法COI基因的約650個“字母”,構成了每一個物種的DNA條碼。
測定COI基因也很簡單,因為它只有幾百個“字母”的長度。這對於現代測序儀器而言簡直易如反掌。而且由於COI基因來自線粒體,它的拷貝數量更多(因為一個細胞只有一個細胞核,卻有許多線粒體)。測定一次COI基因只需要數美元的費用。測出一份樣本中的COI基因的序列之後,把它們與已經測定完成的序列相比較,就可以知道該樣本是否屬於某個已知物種,而不需要利用傳統的分類學方法進行鑑定。
只要把生物體的一小部分———皮、毛髮、葉子等放到DNA條形碼掃瞄器里,地球上每種植物和動物就能通過快速地分析DNA中的片段加以識別,就像在商店裡掃瞄器讀取條形碼那樣。
研究進展
 進展
進展廣東中山檢驗檢疫局參與、由中國檢驗檢疫科學研究院牽頭申報的《檢疫性有害生物DNA條形碼檢測資料庫建設及套用》項目已被科技部列入國家“十二五”科技支撐計畫。
整個項目通過對檢疫性有害生物DNA條形碼檢測技術、標準、資料庫與查詢系統等關鍵技術研究以及綜合示範,突破DNA條形碼檢測技術中不同類群、不同分類單元DNA條形碼篩選與確認,DNA條形碼檢測與現有形態學和分子生物學檢測技術的比對與驗證等技術瓶頸,從而建立我國對外檢疫性有害生物DNA條形碼檢測技術體系,形成DNA條形碼檢測技術標準體系。
DNA條碼技術有助貝類多樣性研究
 DNA條碼用於貝類分類
DNA條碼用於貝類分類簾蛤科是海洋雙殼貝類中物種數量最多的一個門類,命名的種類有500餘種,其中許多種類具有重要經濟價值。然而,由於種內和種間表型的多態性,一些簾蛤科貝類很難單靠形態特徵來鑑定。近年來,人類活動帶來的空前的生物多樣性危機日益嚴重,為了準確評估生物多樣性並進一步有效保護這些形態分類混亂的簾蛤貝類,亟需一種快速而且簡單的方法來界定它們的種的界限。
該研究利用基於線粒體COI基因的DNA條形碼技術,分析了中國沿海60種簾蛤貝類的315個體,發現幾乎所有形態上能夠鑑定到種的個體都形成獨自的DNA條形碼簇;形態上不能確定種名的26個體中,12個體被鑑定到已知種,其餘14個體形成5個單獨的支系,可能代表未描述的新種,或者至少是在國內未記錄的種類。此外,在5個傳統的形態種中發現隱存種的存在。該研究證實了DNA條形碼技術能有效鑑定海洋貝類,同時能通過為分類修訂提供有用信息、指示鑑別性的形態特徵和標示隱存種,輔助分類和評估生物多樣性。這是迄今為止國內外最大規模的海洋貝類DNA條形碼研究。美國洛克菲勒大學MarkStoeckle教授在DNA條形碼部落格中介紹了該研究成果,並對作者首次提出的DNA條形碼除具有物種鑑定和隱存種發掘作用外,還具有“Taxonomydisentanglement”第三種作用的觀點予以充分肯定。
科學家提出的DNA物種識別新方法
 DNA條碼
DNA條碼近日首都師範大學生科院張愛兵教授在著名的進化、生態學國際期刊《分子生態學》(MolecularEcology)上發表了DNA條形碼研究新成果。
張愛兵教授系首都師範大學“海外引進人才”,現為首都師範大學生命科學學院分子生態學學科帶頭人,博士生導師。其領導的研究團隊致力於DNA條形碼理論與套用、有害生物的分子識別和生態安全,目前承擔有國家自然科學基金面上項目等,是國際DNA條形碼理論研究領域少數幾個研究團隊之一,在相關領域具有較強的國際影響力。
DNA條形碼研究是國際生態學、進化生物學領域新興的交叉研究領域(分子生物學,經典分類學,生態學以及計算機科學的交叉),由加拿大科學家P.Hebert於2003年提出,在國際生物學相關領域引起廣泛關注。DNA條形碼研究極大地促進了生物多樣性保護、外來入侵和有害生物分子識別等相關領域的研究。
在這篇文章中,張教授將模糊數學理論引入到DNA條形碼研究領域,提出了基於模糊成員關係與最小遺傳距離的DNA物種識別新方法。他通過實例研究和超過5000次的計算機模擬,證明該方法明顯優於常用的基於Bayesian理論的方法和基於NJ建樹的方法,尤其能夠降低DNA條形碼識別中的假陽性錯誤。
此研究是張愛兵教授繼2008年將人工智慧的思想引入到DNA條形碼(DNAbarcoding)研究領域後,在該領域內取得的又一次理論突破。
將ITS作為種子植物核心DNA條形碼
 不同條形碼和條形碼組合的物種解析度比較
不同條形碼和條形碼組合的物種解析度比較DNA條形碼(DNABarcoding)是利用標準基因片段對物種進行快速和準確鑑定的新技術。加拿大科學家PaulHebert等於2003年提出這一概念後,DNA條形碼已成為生物學領域發展最迅速的學科前沿之一。線粒體細胞色素C氧化酶基因CO1由於其引物通用性高、長度合適、進化速率較快、可以區分近緣物種等優點,已成為較為理想的動物DNA條形碼。隨後,Hebert等人發起了“國際生命條形碼計畫”,並提出了實施方案框架。
由於植物線粒體基因進化速率較慢,CO1基因不能用做植物DNA條形碼。篩選有效的植物DNA條形碼已成為國際生命條形碼研究的首要任務之一。從2005年至今,美、英、韓等國多個研究組提出了若干個植物DNA條形碼或條形碼組合的動議。2009年8月,國際生命條形碼聯盟(CBOL)植物工作組推薦rbcL+matK組合作為陸生植物的DNA條形碼,在同年11月被第三屆國際生命條形碼大會確定為核心條形碼。由於該組合條形碼尚存在研究樣本量偏小和物種解析度低等問題,大會建議trnH-psbA和ITS作為輔助條形碼,並在2011年下半年前對相關條碼進行進一步評價,以最終確定通用的植物DNA條形碼。
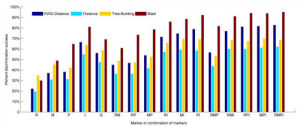 不同條形碼和條形碼組合基於四種分析方法的物種解析度比較
不同條形碼和條形碼組合基於四種分析方法的物種解析度比較作為對國際生命條形碼聯盟這一建議的回應,2009年8月起,在中國科學院大科學裝置開放研究項目“依託種質資源庫的植物DNA條形碼研究”的支持下,中國科學院昆明植物研究所李德銖研究員聯合全國19個科研院所和高校62名研究人員組成的“中國植物條形碼研究團隊(ChinaPlantBOLGroup)”,深入開展了種子植物DNA條形碼的研究。
研究團隊根據對主要來自中國的種子植物75科141屬1757種共約6286個樣本(每個種至少2個樣本)的四個DNA候選條形碼片段(rbcL,matK,trnH-psbA和ITS)引物通用性、序列質量和物種分辯率等的綜合分析,發現三個質體DNA候選條形碼片段具有較高的通用性;核糖體核DNA候選條形碼ITS在被子植物中的通用性較高,而在裸子植物中稍低。研究還發現,ITS具有最高的物種分辯率,與三個質體DNA條形碼片段的任何一個組合均可分辨69.9-79.1%的物種,顯著高於rbcL+matK條形碼組合49.7%的解析度。此外,ITS的部分序列ITS2也表現出較高的物種解析度。
因此,研究團隊建議將ITS作為種子植物的核心DNA條形碼(rbcL+matK+ITS),同時強調了利用多個樣本取樣和不同遺傳方式的DNA片段開展植物DNA條形碼研究的重要性。該文於本月18日線上發表於美國《國家科學院院刊》。
繼2009年由10個國家24個研究單位52名研究人員組成的CBOL植物工作組對7個植物DNA候選條形碼片段評價後,“中國植物條形碼研究團隊”的此項研究是組織規模最大、獲得數據量最豐富和參加人員最多的一項。該文也是該研究團隊2011年5月在“JournalofSystematicsandEvolution”組織發表“中國植物DNA條形碼研究”專輯後又一重要研究成果。
國際生命條形碼聯盟植物工作組主席PeteHollingsworth教授應邀發表專題評論,對中國研究團隊的工作給予了高度評價。他指出,研究團隊通過大樣本取樣的比較分析,對ITS序列的優缺點進行了綜合評估,對將ITS納入植物條形碼的常規運用提供了有說服力的研究案例。他強調,該文的發表代表了將DNA序列數據納入植物物種水平分類和鑑定常規套用邁出的重要一步。
