蛋白質摺疊
在生物體系中,蛋白質摺疊是最重要和最具有挑戰性的問題。隨著計算能力的增強,用全原子分子動力學模擬研究蛋白質摺疊對人們來講已經非常普遍。原則上來講,直接的分子動力學模擬能夠提供蛋白質folding/unfolding的熱動力學信息,但是直接的計算經常受到勢能面上相空間取樣效率問題的阻礙。為了獲得蛋白質構型空間上完全取樣,發展了廣義系統算法包括多重正則算,模擬退火算法和副本交換分子動力學(REMD)方法。在那些方法中,REMD方法是模擬蛋白質摺疊最流行的方法之一。
色氨酸籠
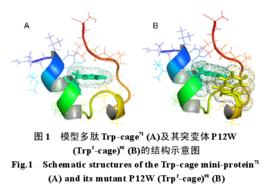
設計具有20個殘基的小蛋白Trp-cage(色氨酸籠),由於它非常小並且很容易摺疊,使它成為架起理論和實驗橋樑的理想的模型。Trp-cage(TC5B:NLYIQWLKDGGPSSGRPPS;PDBentry1L2Y)是由Neidigh等人設計,包括α-螺旋(殘基2-8)310–螺旋(殘基11-14)多聚脯氨酸II螺旋(殘基17-19)。TC5B中疏水核(兩個脯氨酸和3號絡氨酸)被認為是形成摺疊的主要驅動力。摺疊結構被在9號天門冬氨酸和16號精氨酸形成的鹽橋進一步穩定。
Simmerling等人用AMBER99力場和普適的波恩溶劑模型(GB)研究發現在20ns到50ns的動力學模擬中,有一個結構非常靠近NMR結構,重原子的RMSD是1.4Å。Snow等人用OPLS分子力場和GB/SA溶劑模型研究TC5B的摺疊比率並且提出了摺疊時間範圍從1.5μs到8.7μs。Duan等人用AMBER99分子力場和GB模型研究TC5B的快速摺疊機理。從線狀結構出發,他們模擬出最終結構的主鏈原子與NMR結構主鏈原子的RMSD在1.0Å以內。Chen等人用CHARMM22/CMAP全原子分子力場和GBSW隱式溶劑模型成功摺疊了TC5B。
儘管TC5B的NMR結構已經被各種各樣的計算模擬正確預測,但是對TC5B的folding/unfolding的動力學行為的理論模擬並沒有正確的結論。Pitera等人用AMBER94分子力場和GB/SA溶劑模型進行REMD模擬來研究TC5B的摺疊行為,他們計算的熔解溫度大約400K,遠遠高於實驗值315K。Zhou等人用OPLA-AA分子力場和顯式溶劑模型來進行REMD模擬並且發現熔解溫度在440K左右。Paschek等人發現摺疊的TC5B是太穩定,熔解溫度高出實驗值130K。上述那些結果,在不同的分子力場和不同的溶劑模型下,都顯示了比實驗值更高的熔解溫度。注意上述的那些結果經常依靠分子力場以及所套用的溶劑模型。Shell等人最近研究表明:蛋白質的穩定性對所用的分子力場和的溶劑模型是非常敏感的 。
色氨酸籠摺疊形成機制的研究進展
色氨酸籠的摺疊過渡溫度
目前國內外科研工作者通過核磁共振(NMR)、圓二色(CD)光譜、螢光猝滅光譜(FQCTS)、紫外共振拉曼光譜(URRS)等實驗手段對色氨酸籠的摺疊機制都已進行過深入的研究,結果表明其摺疊過程可在1.0-4.1μs範圍內完成,該結果也得到了理論模擬計算的驗證。然而,目前對於色氨酸籠的一些具體的摺疊細節過程仍然存在很大爭議和疑問。例如Qiu等通過跳溫波譜技術(TJS)研究表明色氨酸籠通過兩態協同的方式完成摺疊過程;但Neuweiler和Ahmed課題組發現色氨酸籠並非只是一個簡單的兩態摺疊蛋白;還有一些課題組利用分子動力學模擬方法對Trp-cage的摺疊路徑和機理進行研究發現:Trp-cage摺疊轉變過程中過渡態形成的溫度約為400K或更高溫度,未能重複得出實驗所得到的過渡態的溫度(315K);而Day、Duan和Zhang等對Trp-cage摺疊過程進行的副本交換分子動力學(REMD)模擬發現,溶解溫度可以和實驗值相吻合。通過分析比較可以推測,這些模擬計算結果的差異性主要是由於動力學模擬所採用分子力場與溶劑模型的不同。基於這種模擬計算和實驗研究結果的不一致,最近O-dziej等採用二維核磁共振技術與分子動力學模擬相結合的方法對Trp-cage的摺疊過渡溫度進行了研究,結果表明:由於色氨酸籠摺疊複雜性(關鍵胺基酸Trp6和Pro12的疏水長程協同作用與N端α-helix解旋過程同時發生),其摺疊過渡溫度並非在某一溫度時刻,而是在311-317K範圍內。
色氨酸籠的摺疊形成機制
目前對於色氨酸籠摺疊過程中各個摺疊細節發生的順序一直都未達成共識。例如國內外一些研究蛋白摺疊課題組發現鹽橋Asp9/Arg16的率先形成對色氨酸籠的摺疊發揮著至關重要的作用,然而這恰恰與實驗得到的結果相反。通過圓二色譜實驗研究發現,將鹽橋Asp9/Arg16去掉後,色氨酸籠的摺疊時間未發生明顯的變化,並且最近研究也發現模擬計算起始構象中的鹽橋Asp9/Arg16甚至並不能穩定存在,在模擬過程中會發生斷裂消失,並且N端α-helix的穩定存在與其形成具有緊密聯繫。Chowdhury和Hu等運用全原子分子動力學模擬發現疏水核心Trp6的包埋掩藏是色氨酸籠摺疊過程的關鍵限速步驟,而Juraszek等採用副本交換分子動力學模擬發現Trp-cage摺疊過程的限速步驟並非絕對為疏水核心吲哚環的包埋,其研究結果表明色氨酸籠N-端上的α-螺旋結構形成於摺疊過程的早期,之後疏水殘基逐漸向疏水核心(Trp6)靠攏,最終疏水核中的Trp6側鏈上的吲哚環完整包埋而結束整個摺疊過程。Mok等通過脈衝光譜實驗研究發現在非摺疊態結構的Trp-cage中存在疏水核結構,說明疏水核的塌陷推動蛋白結構的整個摺疊進程,即所謂的疏水塌陷模型。Trp-cage這種疏水塌陷摺疊模型的提出也得到了其他課題組的支持,而Ahmed課題組發現結構中N端的α-螺旋結構的形成先於疏水核的塌陷,Juraszek課題組卻認為Trp-cage的摺疊形成遵循成核-聚集和擴散-碰撞模型。可見,雖然色氨酸籠只有20個胺基酸組成,且其結構簡單緊湊且摺疊迅速,但其摺疊機制仍然存在爭議。為了探究該色氨酸籠複雜的摺疊過程,我們曾採用溫控分子動力學(TCMD)方法對色氨酸籠在真空和水溶劑條件下的摺疊與解摺疊過程進行了深入的研究,得到了Trp-cage摺疊過程中的重要中間態結構,為其摺疊與解摺疊機制的揭示提供了直接、可靠的依據;結果發現溶劑水分子可通過疏水作用誘導Trp-cage形成一個亞穩態(MSOL)和一個非天然態疏水核的過渡態(TS)結構,推動肽鏈疏水殘基間的相互作用,進而促進肽鏈結構疏水核的快速收縮和正確摺疊。為深入理解Trp-cage天然摺疊態構型的穩定形成機制,我們採用分子動力學結合殘基點突變(甘氨酸替換)的方法研究Trp-cage中各個胺基酸側鏈基團在其結構穩定性中的作用,通過與野生天然態蛋白結構穩定性特徵數據對比,確定了每個殘基側鏈在其整個結構穩定性中的作用。如殘基Tyr3與Pro19間的疏水作用、殘基Ser14和Arg16間的氫鍵作用以及Pro17所形成的三級天然接觸等穩定作用對Trp-cage整體結構的穩定性起著特殊重要的作用。另外,具有相同側鏈的胺基酸殘基(Ser13和Ser14)由於其側鏈所處位置的不同導致其對結構的穩定性的貢獻程度也不同;鹽橋在模型多肽結構中本身穩定性非常高,不易被突變所破壞。這些結果為理解色氨酸籠摺疊構型的穩定形成機制提供了重要的理論依據 。